Notice
Présentation des performances paralléles du code QDD CUDA Fortran sur différentes Architectures GPU
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
QDD est l'acronyme de Quantum Dissipative Dynamics, un ensemble de théories développées pour prendre en compte les corrélations dynamiques incohérentes dans les clusters et les molécules. Les corrélations dynamiques dépassent la dynamique de champ moyen et deviennent prédominantes dans les situations très éloignées de l'équilibre, avec de fortes énergies d'excitation, et sont responsables des comportements dissipatifs. En raison de la nécessité d'une grande précision pour capturer les effets hautement non linéaires, la grille de simulation doit avoir une résolution élevée, ce qui rend le calcul de la dynamique coûteux en termes de temps.
Le rendement de cette nouvelle implémentation sur GPU a été testé et comparé à la parallélisation OpenMP sur de petits clusters de sodium et de petites molécules covalentes. La parallélisation OpenMP permet un gain de vitesse moyen d'un ordre de grandeur par rapport à un calcul séquentiel. L'utilisation d'un GPU permet un gain supplémentaire d'un autre ordre de grandeur. Une stratégie de minimisation des communications entre le CPU et le GPU, a été implémentée. Cette stratégie peut conduire à des choix de portage de routines du code sur GPU de manière contre-intuitive par rapport aux typologies algorithmiques sous-jaçentes. Cependant la rapport perte/gain sur la globalité de l'exécution du code s'avère largement en faveur du gain d'accélération. Afin de tester la robustesse de l'approche cette Stratégie a été testée sur différentes Architecture GPU, avec différents débits CPUGPU, et différentes Architecures CPU (x86 vs. ARM).
Ce travail de parallélisation sur GPU ouvre la voie à des applications qui étaient auparavant inaccessibles. Grâce à l'utilisation des capacités de calcul du GPU, une accélération significative a été obtenue, ouvrant de nouvelles opportunités pour des recherches futures dans ce domaine.
Intervention / Responsable scientifique
Thème
Documentation
Dans la même collection
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 2 : Les ateliers de la données
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 7 : Conclusion
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 4 : Des interactions croisées, liens entre le calcul et les données.
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 1 : Introduction et présentation des intervenants
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 6 : L'accompagnement autour de la gestion des données
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 3 : les liens entre les structures
-
Table ronde et discussions : infrastructures de calcul et ateliers de la donnée de recherche Data G…
CastexStéphanieRenardArnaudAlbaretLucieRenonNicolasDufayardJean-FrançoisPARTIE 5 : Des interactions croisées, à propos des compétences
-
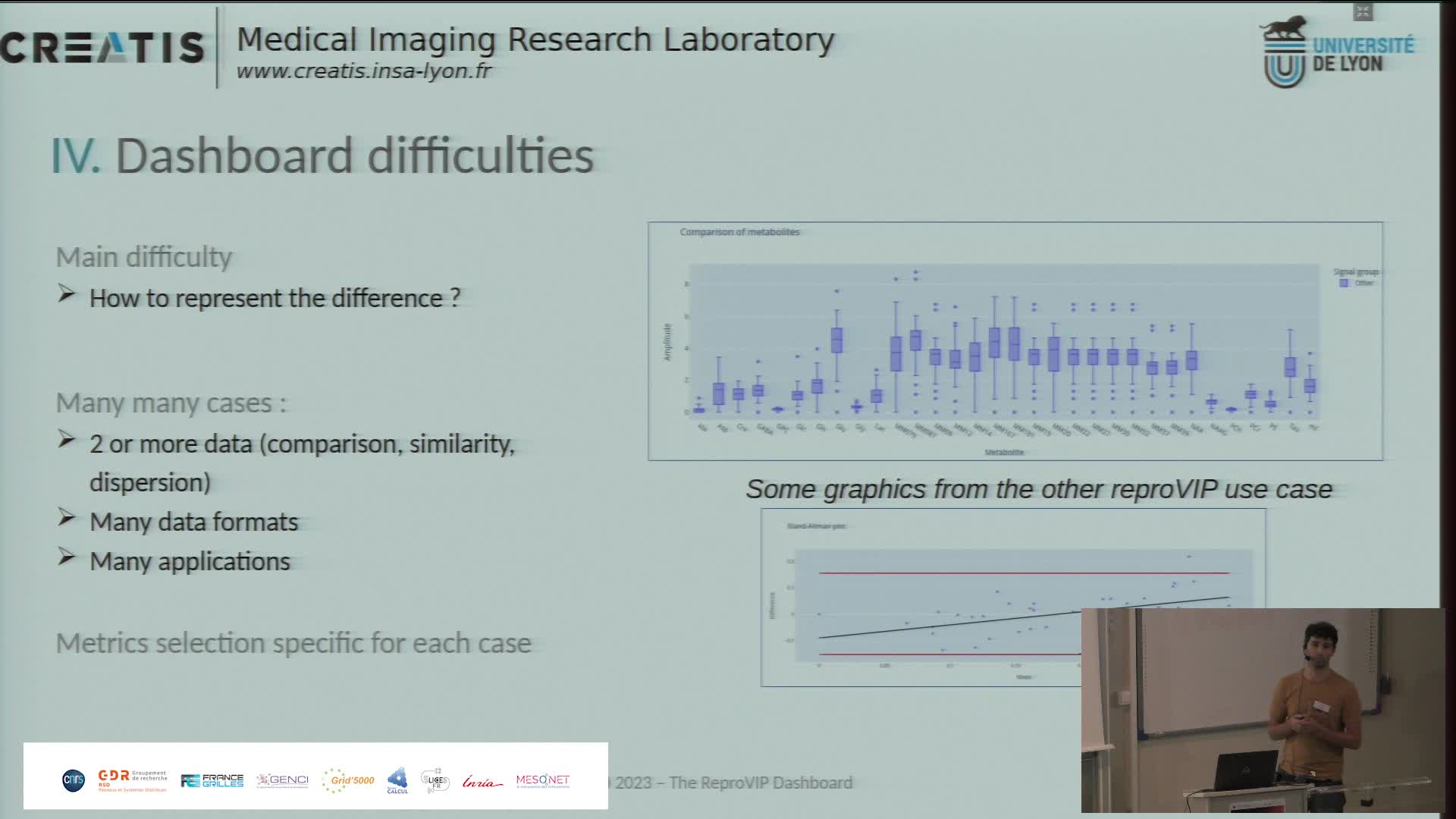
Présentation du dashboard ReproVIP pour visualiser la reproductibilité dans l'imagerie médicale
BonnetAxelLa plateforme d'imagerie virtuelle VIP [1] (https://vip.creatis.insa-lyon.fr) est un portail web de simulation et d'analyse d'images médicales. Elle existe depuis plus de 10 ans et a évolué pour
-
Parallélisation par l'intermédiaire d'une fenêtre à mémoire partagée (MPI 3.0) : application à un c…
ElyakimePierreJADIM est un code de calcul de mécanique des fluides développé en Fortran 90 à l'Institut de Mécanique des Fluides de Toulouse (IMFT).
-
Fast Polynomial Evaluation (présentation + demo)
VigneronFrançoisWe propose a new algorithm for quickly evaluating polynomials. The FPE algorithm pre-conditions a complex polynomial P of degree d in time O(d log d), with a low multiplicative constant independent
-
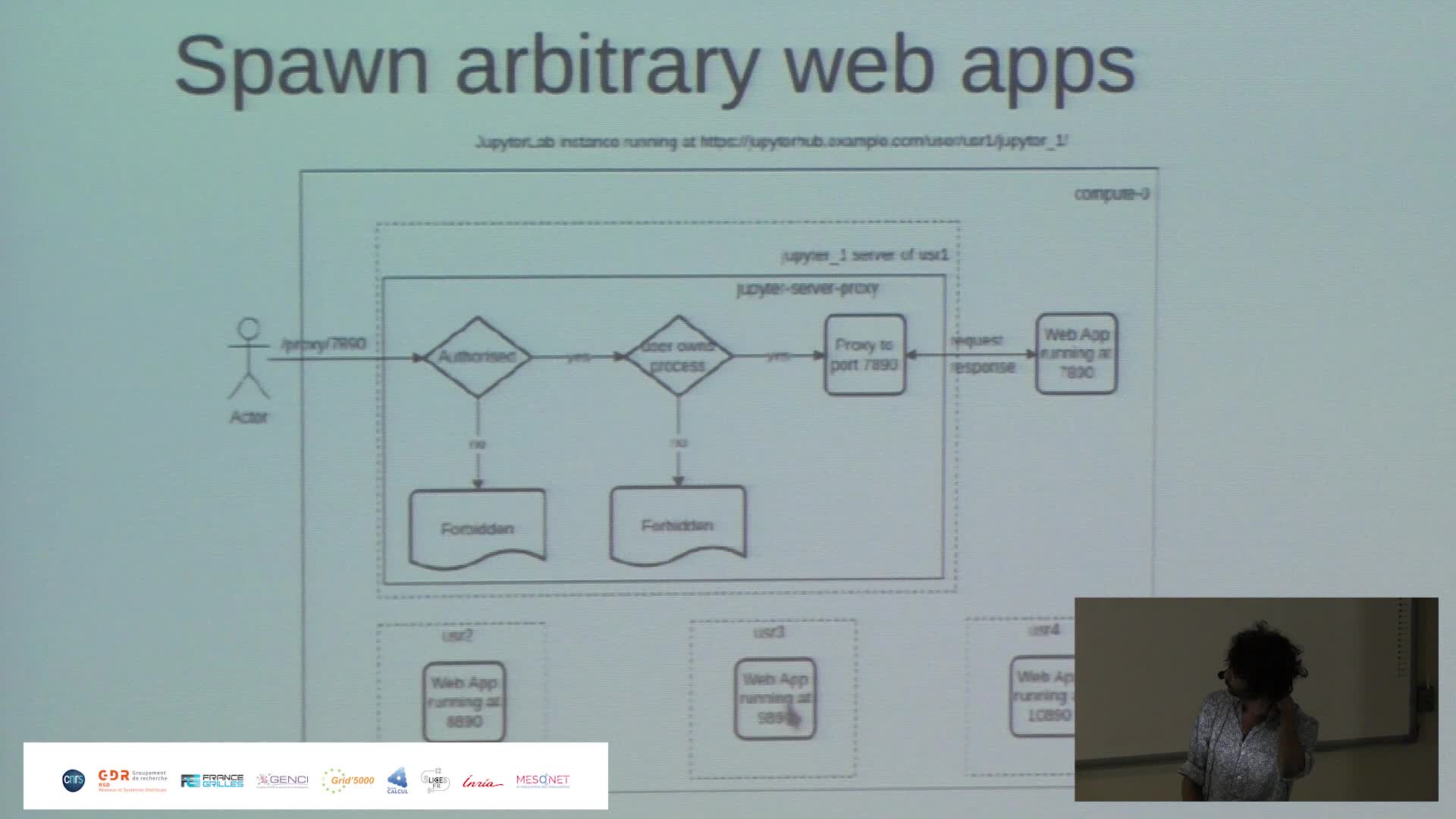
Harnessing the power of Jupyter{Hub,Lab} to make Jean Zay HPC resources more accessible
PaipuriMahendraThis talk revolves around the deployment of JupyterHub on Jean Zay HPC platform and how it is done to meet the demands of RSSI (ZRR constraints) and also to provide a seamless experience to the end
-
Using High Performance Computing to decipher the impact of ageing process on collagens
DepenveillerCamillePlant cells have to face environmental stress, and in this context, being able to decipher the organization of the plant plasma membrane is a key point to better understand this adaptability of plasma
Avec les mêmes intervenants et intervenantes
-
Harnessing the power of Jupyter{Hub,Lab} to make Jean Zay HPC resources more accessible
PaipuriMahendraThis talk revolves around the deployment of JupyterHub on Jean Zay HPC platform and how it is done to meet the demands of RSSI (ZRR constraints) and also to provide a seamless experience to the end