Notice
Parallelization des methodes Particle-In-Cell avec reconstructions sur grilles parcimonieuses (sparse grids), application à la physique des plasmas
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
Basée sur une discrétisation mixte, composée d'un maillage et de particules, du système de Vlasov-Poisson, la méthode Particle-In-Cell est parmi les plus populaires dans le domaine de la simulation numérique des plasmas cinétiques. Cette méthode présente plusieurs avantages: sa robustesse, sa simplicité d'implémentation ou encore sa facilité de parallélisation sur plusieurs nœuds de calcul. Néanmoins, à cause d'une représentation statistique de certaines quantités, un nombre important de particules est nécessaire pour maintenir un niveau de bruit numérique acceptable, ce nombre pouvant atteindre 10^{11} particules dans le cas de certaines simulations tri-dimensionnelles. Actuellement, cette contrainte limite la plupart des simulations à des modèles réduits en deux dimensions et constitue le principal frein à l'extension aux problèmes tri-dimensionnels. En conséquence, la question du stockage ainsi que de l'accès de ces données de manière efficace, en séquentiel ou en parallèle, est un challenge important qui a suscité un intérêt soutenu ces dernières années.
Récemment, une technique d'interpolation basée sur une reconstruction de la solution à partir de grilles parcimonieuses (sparse grids) a été appliquée à la méthode, dans le but de réduire drastiquement le coût en mémoire de la méthode, ainsi que les temps de calcul. En effet, après avoir étudié certaines propriétés de convergence et de stabilité de la méthode [1], nous avons montré que pour les simulations les plus coûteuses, la quantité de données à traiter peut être réduite jusqu'à quatre ordre de grandeur et le temps de calcul jusqu'à deux ordres de grandeur [2].
Au cours de cette présentation nous proposons d'aborder dans un premier temps la parallélisation de cette méthode dans un contexte de mémoire partagée (OpenMP), puis dans un second temps le portage de la méthode sur accélérateur graphique (GPGPU). Ces deux types de parallélisation représentent un challenge important au vue de la difficulté d'obtenir une stratégie efficace dans le cas des méthodes PIC classiques à cause de la quantité de données à gérer (code memory-bound, données dépassant la mémoire d'un nœud GPU et nécessité de transfert entre host et device, etc.)
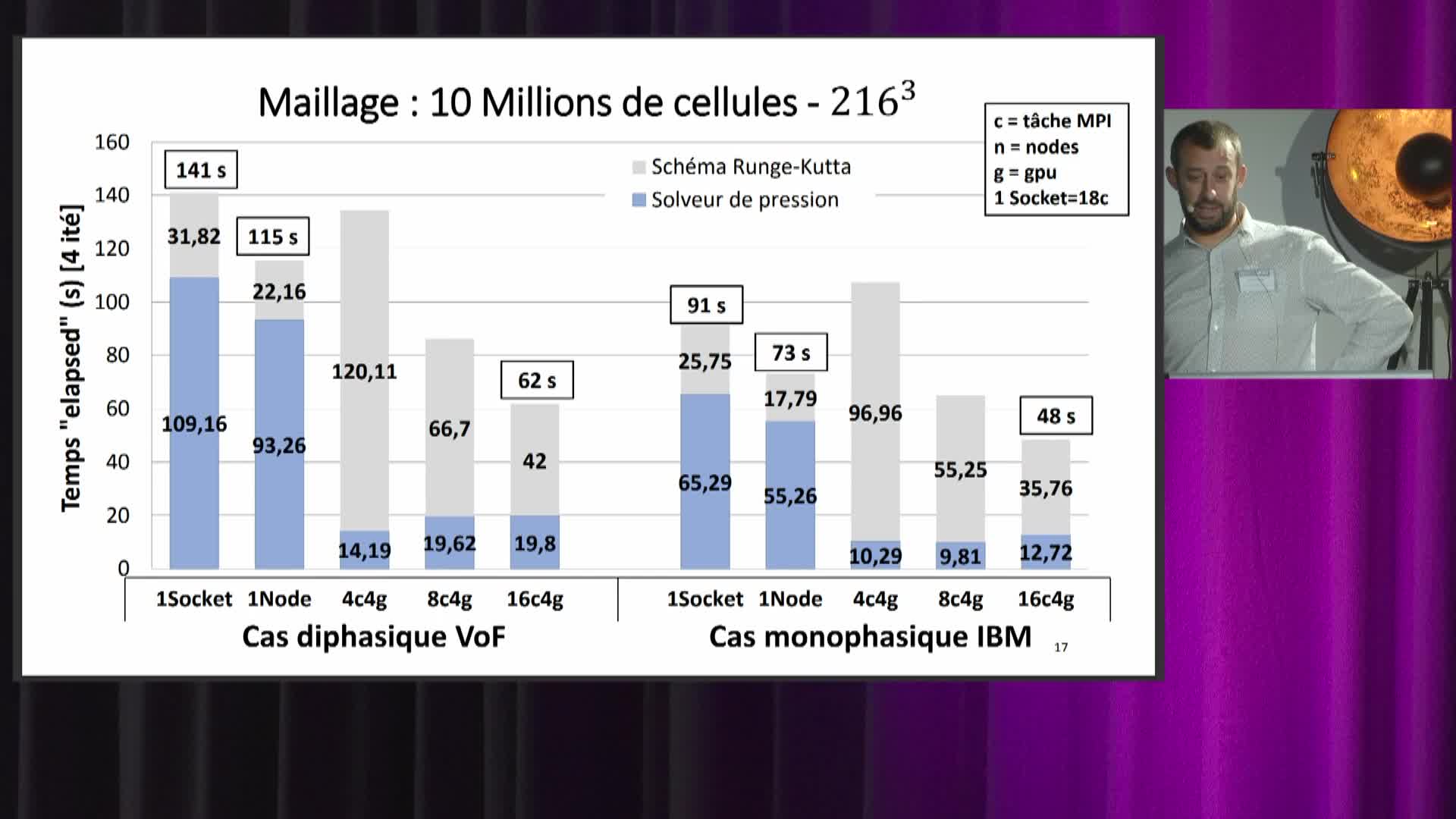
Grâce à l'introduction de nouvelles stratégies de parallélisation (basées sur les particules et les différentes grilles intervenant dans la méthode), d'une réutilisation optimale du premier niveau de mémoire cache et d'un équilibrage des charges de travail nous avons obtenu un speedup quasi-parfait sur l'étape qui est de loin la plus coûteuse (90\% du temps de calcul) de l'algorithme (126 pour 128 cœurs, speedup de 101 sur l'algorithme complet) pour une architecture Non-Uniform Memory Access (NUMA) à mémoire partagée (Two processor sockets AMD EPYC 7713 Milan), ainsi que pour une architecture à mémoire uniforme de huit cœurs (Intel Core i9-10885H).
Nous avons également proposé deux algorithmes alternatifs de la méthode, basé sur l'architecture des GPUs et exploitant la forte capacité de calcul de ces derniers permettant d'obtenir un speedup six fois plus important que notre implémentation naïve de la méthode sur GPU (Nvidia Tesla GV100).
L'originalité des travaux repose sur le caractère novateur de la parallélisation de la méthode sparse PIC, qui n'a jamais été abordée à notre connaissance, ainsi que des stratégies de parallélisation proposées.
[1] F. Deluzet, G. Fubiani, L. Garrigues, C. Guillet, and J. Narski. Sparse grid reconstructions for particle-in-cell methods. Submitted, 2022.
[2] F. Deluzet, G. Fubiani, L. Garrigues, C. Guillet, and J. Narski. Efficient shared memory parallelization for sparse grid Particle-In-Cell. Submitted, 2022.
Thème
Documentation
Dans la même collection
-
-
-
-
-
-
-
-
-
Recherche Data Gouv : un écosystème au service du partage et de l'ouverture des données de recherche
Un écosystème au service du partage et de l’ouverture des données de recherche
-
-
-
Refroidissement par immersion de serveurs : premiers retours opérationnels
Le refroidissement par immersion n'est pas une nouveauté, de l'IT en général au HPC en particulier. Il y a 35 ans, les Cray-2 et successeurs avaient leur assemblage de tours immergées dans un fluide