Notice
ADELF-EMOIS 2012 – Interopérabilité des bases de données médicales : proposition d’une méthode de mise en correspondance des bases de biologie optimisant leur exploitation.
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
Titre : ADELF-EMOIS 2012 – Interopérabilité des bases de données médicales : proposition d’une méthode de mise en correspondance des bases de biologie optimisant leur exploitation.
Auteurs : Grégoire FICHEUR (EA2694, Université Lille Nord de France ; France)
Jean-Baptiste BEUSCART (EA2694, Université Lille Nord de France ; France)
Aurélien SCHAFFAR (EA2694, Université Lille Nord de France ; France)
Emmanuel CHAZARD (EA2694, Université Lille Nord de France ; France)
Résumé : Introduction
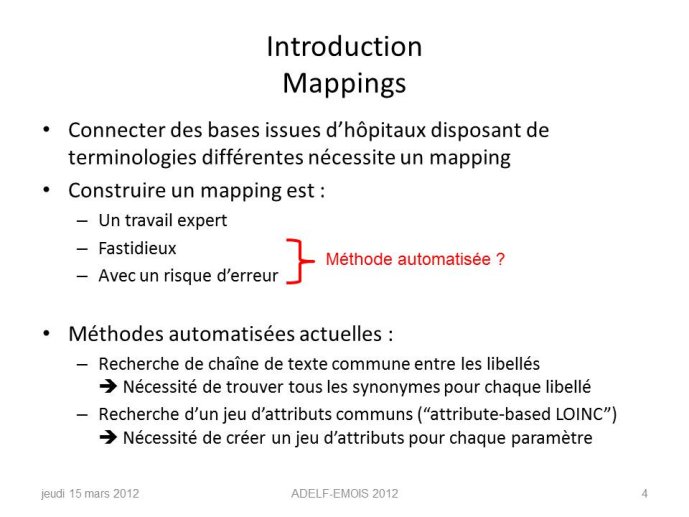
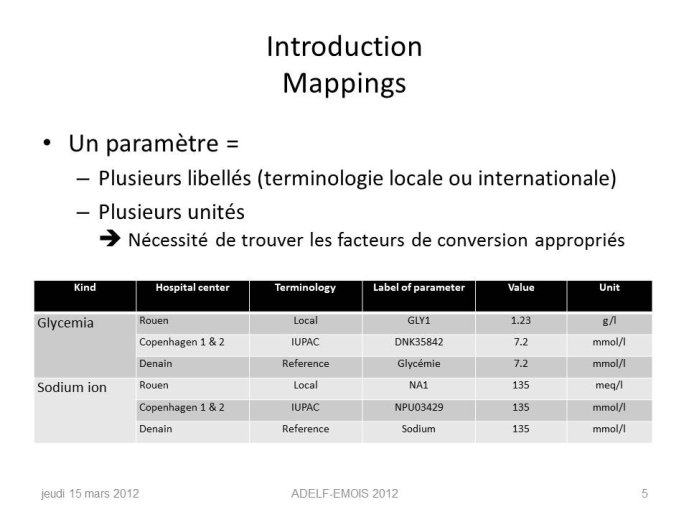
L'utilisation des résultats de biologie est intéressante en recherche, et également en contrôle de codage du PMSI (exemple : malnutritions, dysnatrémies, etc.). Cependant, dans les systèmes d'information hospitaliers, les résultats de biologie sont stockés en utilisant des terminologies locales ce qui rend difficile l'identification des paramètres pertinents devant être utilisés pour ce contrôle. L'objectif est de créer un outil aidant à construire un mapping entre une terminologie de référence contenant les paramètres d'intérêt et une terminologie locale.
Méthodes
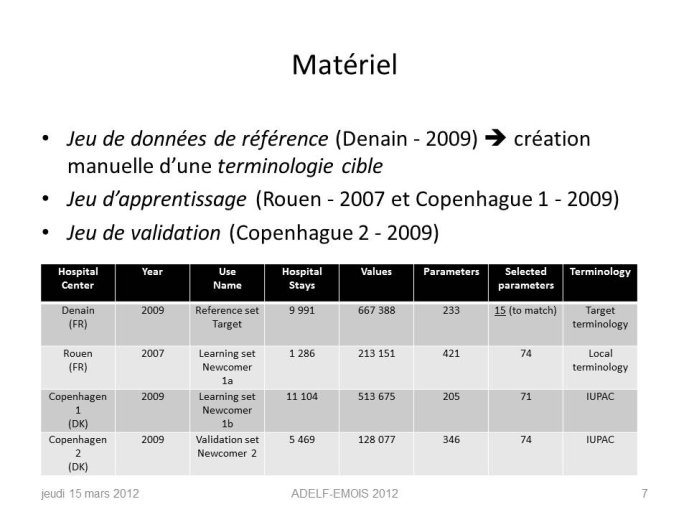
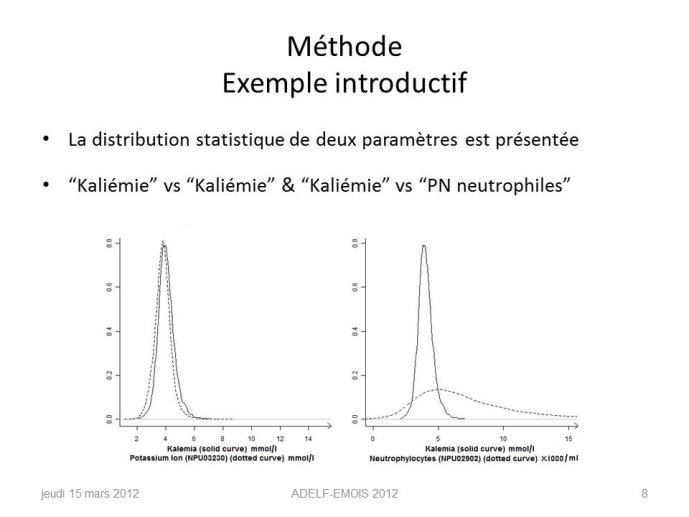
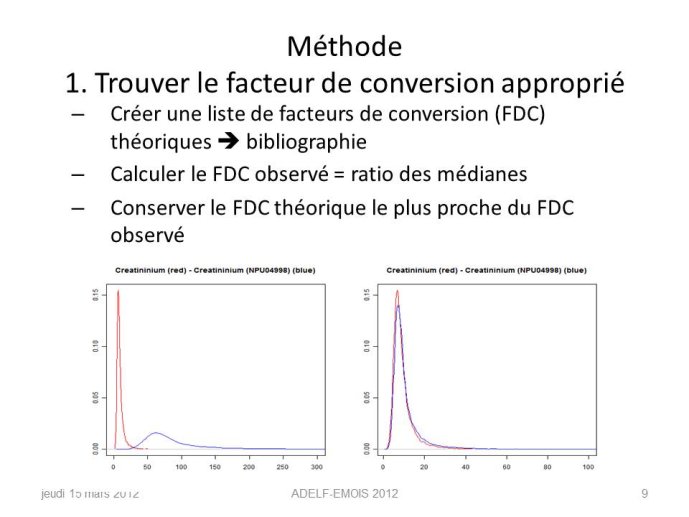
En utilisant un échantillon d'apprentissage constitué de correspondances correctes et incorrectes entre les paramètres de différents hôpitaux, un score de probabilité de correspondance est construit, il repose sur la comparaison des distributions statistiques des paramètres de biologie. Ce modèle permet également de déterminer le facteur de conversion entre paramètres d’unités différentes. Cette méthode est évaluée sur un échantillon de test d'un nouvel hôpital : pour chaque paramètre de référence, les meilleurs candidats sont retournés et triés par ordre décroissant en utilisant ce score.
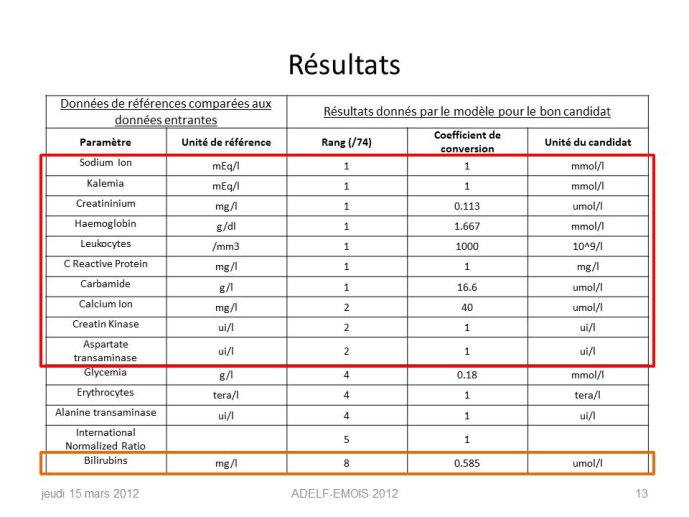
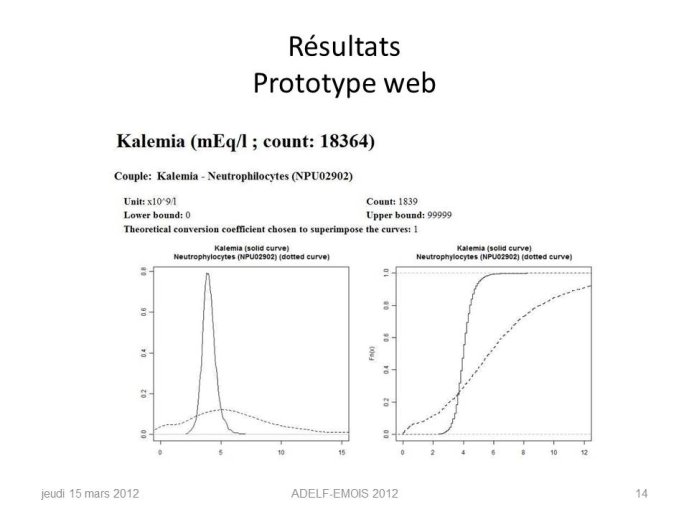
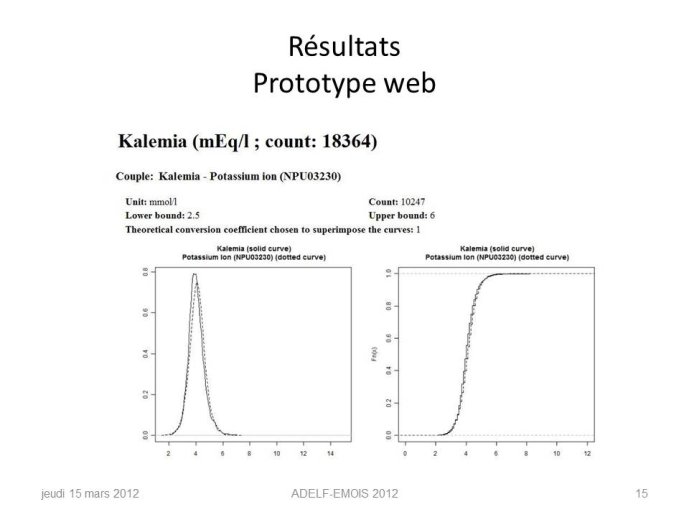
Résultats
Parmi plus de 70 paramètres, le bon paramètre est retrouvé dans le top 5 des propositions automatiques dans 14 cas sur 15. Tous les facteurs de conversion sont corrects. Un outil web présente les informations essentielles concernant les meilleurs candidats. Grâce à cet outil, un expert a trouvé tous les paramètres pertinents dans la base du nouvel hôpital.
Discussion/conclusion
Le fait de présenter sur un outil web une liste courte de paramètres candidats (donnée par le modèle pour un paramètre de référence) apporte une aide essentielle dans l'identification des paramètres pertinents pouvant être utilisés dans la chaîne de contrôle du codage de l'activité. Cet outil semble constituer une aide précieuse pour les départements d'information médicale désirant exploiter les résultats de biologie dans cette perspective.
L’auteur n’a pas transmis de conflit d’intérêt concernant les données diffusées dans cette vidéo ou publiées dans la référence citée.
Conférence enregistrée lors du IIIème Congrès National conjoint ADELF/EMOIS à Dijon les 12 et 13 mars 2012. Session : Systèmes d’informations (Salle Morey Saint-Denis). Modérateurs : F KOHLER, E SAULEAU.
Réalisation, production : Canal U/3S - CERIMES
Mots clés : ADELF, EMOIS, 2012, Dijon, épidémiologie, langue française, évaluation, management, organisation, information, santé, PMSI, Interopérabilité, CIM-10, biologie
Intervention
Thème
Documentation
Dans la même collection
-
ADELF-EMOIS 2012 – Présentation du projet FIDES (Facturation individuelle des établissements de san…
PonsPierre-CharlesRobinFrankTitre : ADELF-EMOIS 2012 – Présentation du projet FIDES (Facturation individuelle des établissements de santé) Auteurs : Yannick LE GUEN (sous-directeur de la performance des acteurs de l’offre de
-
ADELF-EMOIS 2012 – Atelier : les différents outils d'information.
BensadonMaxTitre : ADELF-EMOIS 2012 – Atelier : les différents outils d'information.Animé par l'ATIHAuteurs : Max BENSADON (Directeur adjoint de l'ATIH).Résumé : Présentations de l’ATIH (Agence technique de l
-
ADELF-EMOIS 2012 – Projet T2A SSR : État d’avancement des travaux.
BuronfosseAnneSchererAntoinetteDubois-LefrèreJoëlleDucretNathalieTitre : ADELF-EMOIS 2012 – Projet T2A SSR : État d’avancement des travaux.Auteurs : BURONFOSSE Anne (Santé publique et médecine sociale), DUCRET N, DUBOIS J, SCHERER A.Résumé : Présentations de l’ATIH
-
ADELF-EMOIS 2012 - les dernières évolutions : CMD 14 et 15.
Cadavid GomezSandraTitre : ADELF-EMOIS 2012 - les dernières évolutions : CMD 14 et 15...Auteurs : GOMEZ Sandra (ATIH), BRAMI Michèle (MCO, ATIH).Résumé : Présentations de l’ATIH (Agence technique de l’information sur l
-
ADELF-EMOIS 2012 - Comparaison du diagnostic principal de la dernière hospitalisation et de la caus…
Mechinaud Lamarche - VadelAgatheTitre : ADELF-EMOIS 2012 - Comparaison du diagnostic principal de la dernière hospitalisation et de la cause initiale du certificat de décès en 2008-2009, France.Auteurs : Agathe Lamarche-Vadel
-
ADELF-EMOIS 2012 - Allocution d’ouverture du congrès.
QuantinCatherineTitre : ADELF-EMOIS 2012 - Allocution d’ouverture du congrès.Intervenant : Catherine QUANTIN (Dijon).Résumé : ADELF-EMOIS 2012 - Allocution d’ouverture du congrès. Rencontres organisées conjointement
-
ADELF-EMOIS 2012 – La sécurité et confidentialité des données de santé lors de leur réutilisation à…
GarriguesBernardQuantinCatherineTitre : ADELF-EMOIS 2012 – La sécurité et confidentialité des données de santé lors de leur réutilisation à des fins de traitement statistiqueAuteurs : Table ronde : Interventions de représentants de
-
ADELF-EMOIS 2012 – Projet FIDES (Facturation individuelle des établissements de santé). Retour d'ex…
CharbitHervéScellierMarie-PierreRagainDidierTitre : ADELF-EMOIS 2012 – Projet FIDES (Facturation individuelle des établissements de santé). Retour d'expérience d'hôpitaux.Auteurs : CHARBIT Hervé, LABBE Joëlle, BARREAUX Micheline, OTTON
-
ADELF-EMOIS 2012 – Pharmaco épidémiologie vaccinale pédiatrique : validité de l’identification des …
QuantinCatherineTitre : ADELF-EMOIS 2012 – Pharmaco épidémiologie vaccinale pédiatrique : validité de l’identification des cas de convulsions fébriles à partir du programme de médicalisation des systèmes d
-
ADELF-EMOIS 2012 - Évaluation des ratios normalisés de mortalité hospitalière à partir des données …
LefebvreYannickTitre : ADELF-EMOIS 2012 - Évaluation des ratios normalisés de mortalité hospitalière à partir des données PMSI : comparaison de modèles d’ajustement. Auteurs : Yannick Lefebvre (Agence Technique de l
-
ADELF-EMOIS 2012 – Détection du début de prise en charge hospitalière des patients atteints d'un ca…
TrombertBéatriceTitre : ADELF-EMOIS 2012 – Détection du début de prise en charge hospitalière des patients atteints d’un cancer colorectal dans les bases PMSI de quatre établissements Rhône-alpins.Auteurs : BEATRICE
-
ADELF-EMOIS 2012 - Discussion sur la mortalité : épidémiologie et confidentialité. Session A1
LasbeurLindaLegleyeStéphaneMechinaud Lamarche - VadelAgatheLefebvreYannickTitre : ADELF-EMOIS 2012 - Discussion sur la mortalité : épidémiologie et confidentialité. Session A1 Auteurs : Yannick Lefebvre (Agence Technique de l'Information sur l'Hospitalisation). Agathe
Avec les mêmes intervenants et intervenantes
-
Journées EMOIS Nancy 2015 : Session D2 - Réutilisation du fichier FichComp de la base nationale du …
MetralPierreFicheurGrégoireColinCyrilleJournées EMOIS Nancy 2015 - Session D2 - Réutilisation du fichier FichComp de la base nationale du PMSI pour explorer les complications mécaniques des prothèses totales de hanche – Auditorium 300
-
Journées EMOIS Nancy 2015 : Session B2 - EpiHosp : un outil web permettant l’exploration des poses …
FicheurGrégoireChazardEmmanuelJayNicolasTitre : Journées EMOIS Nancy 2015 - Session B2 - EpiHosp : un outil web permettant l’exploration des poses de dispositifs médicaux implantables et de leurs complications par la réutilisation de la
-
Journées Emois Paris 2014: Codage automatisé à partir des courriers hospitaliers, des résultats de …
FicheurGrégoireTitre : Journées Emois Paris 2014: Codage automatisé à partir des courriers hospitaliers, des résultats de biologie et des prescriptions médicamenteuses : construction automatisée et évaluation de
-
Journées EMOIS Nancy 2013 : Proposition d’une méthode automatisée calculant la valeur moyenne d’un …
FicheurGrégoireG-1-5 Titre : Journées EMOIS Nancy 2013 : Proposition d’une méthode automatisée calculant la valeur moyenne d’un diagnostic associé significatif. Auteur (s) : Grégoire FICHEUR (EA2694, Université
-
EMOIS Nancy 2011 - Codage automatisé : ontologie médicale construite par fouille de textes.
FicheurGrégoireTitre : Codage automatisé : proposition d’une méthode utilisant une ontologie médicale construite par fouille de textes. Résumé : Le codage automatisé est devenu un enjeu médico-économique majeur.Deux