Notice
Retour d'expérience sur le développement d'un cas d'usage en bio-informatique au sein du projet EOSC-Pillar
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
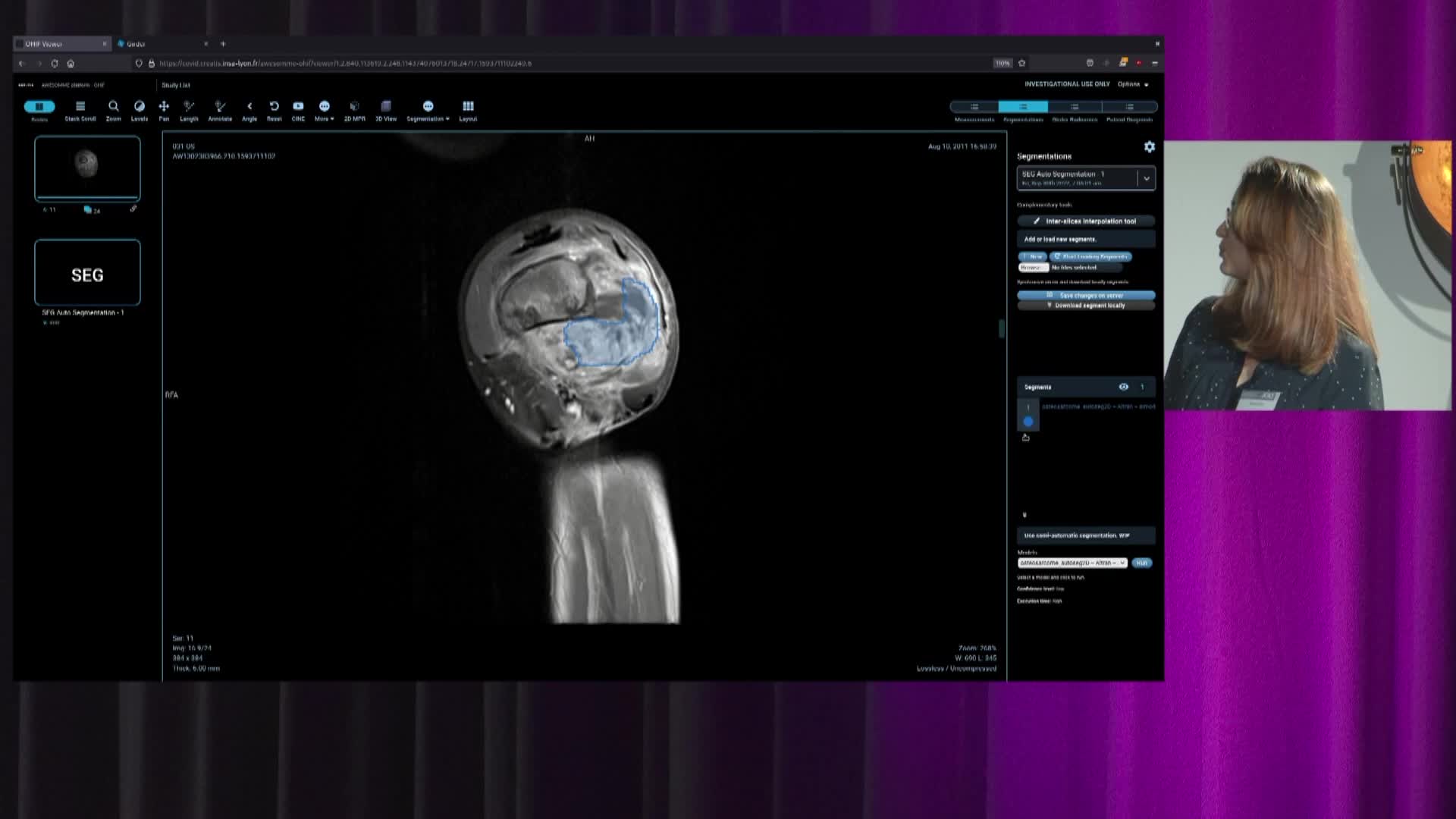
Galaxy est une plateforme de gestion de workflow très utilisée en bioinformatique, et dont l'objectif est de rendre la biologie computationnelle accessible aux chercheurs et scientifiques n'ayant pas ou peu d'expérience de programmation ou d'administration système.
Dans le cadre du projet européen EOSC-Pillar, nous avons construit un cas d'usage scientifique pour répondre à plusieurs questions techniques et organisationnelle liée à l'utilisation de Galaxy : comment connecter cette plateforme avec différentes sources de données ? Comment garantir la reproductibilité des expériences lorsqu'on utilise différentes instances de l'outil ? Peut-on l'utiliser pour traiter des données de santé hébergées sur une plateforme sécurisée ?
Nous proposons, au cours de la présentation, de détailler la façon dont nous avons tenté de répondre à ces questions dans le cadre du projet, en répondant aux quatre objectifs suivants :
- Permettre pour tous les utilisateurs EOSC l'accès à des données de référence depuis différents modes de déploiement de Galaxy ;
- Faciliter le déploiement d'instances de Galaxy sur les infrastructures où sont déposées les données, pour une analyse locale ;
- Apporter une coherence globale dans le déploiement de différentes instances de Galaxy
- Prendre en compte les contraintes liées au traitement de données sensibles (en particulier les données de santé)
Nous décrirons les scénarios théoriques imaginés pour couvrir les différents cas d'usage souhaités. Nous présenterons les services techniques du projet EOSC-Pillar sur lesquels s'appuie notre implémentation, notamment la plateforme Laniakea fournie par les instituts italiens INFN et CNR-IBIOM. La présentation décrira également comment les données sources sont référencée et collectée en utilisant un autre service d'EOSC-Pillar, le FAIR Federated Data Space (F2DS), et comment il est prévu de les consommer à l'autre bout, en interfaçant cet espace avec Laniakea.
Nous donnerons également un exemple concret d'utilisation, en nous basant sur les travaux de la communauté hCNV d'Elixir, pour un travail de benchmarking d'outils d'analyse.
Nous terminerons la présentation par un échange d'impression sur l'intérêt d'outils développés au niveau d'EOSC, et les perspectives d'évolution des services proposés sur la base de ces outils.
Références :
- Galaxy Community Hub : https://galaxyproject.org/
- EOSC-Pillar : https://eosc-pillar.eu/
- EOSC-Pillar T6.6 usecase: https://eosc-pillar.eu/use-cases/exploring-reference-data-through-existing-computing-services-bioinformatics-community
- EOSC Pillar Federated Data Space : https://eosc-pillar.eu/establishing-fair-data-services
Elixir hCNV community : https://elixir-europe.org/communities/hcnv
Thème
Documentation
Dans la même collection
-
-
-
-
-
-
-
-
Recherche Data Gouv : un écosystème au service du partage et de l'ouverture des données de recherche
Un écosystème au service du partage et de l’ouverture des données de recherche
-
-
-
Refroidissement par immersion de serveurs : premiers retours opérationnels
Le refroidissement par immersion n'est pas une nouveauté, de l'IT en général au HPC en particulier. Il y a 35 ans, les Cray-2 et successeurs avaient leur assemblage de tours immergées dans un fluide
-