Notice
Projet ReproVIP : Vers des Résultats Reproductibles en Imagerie Médicale
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
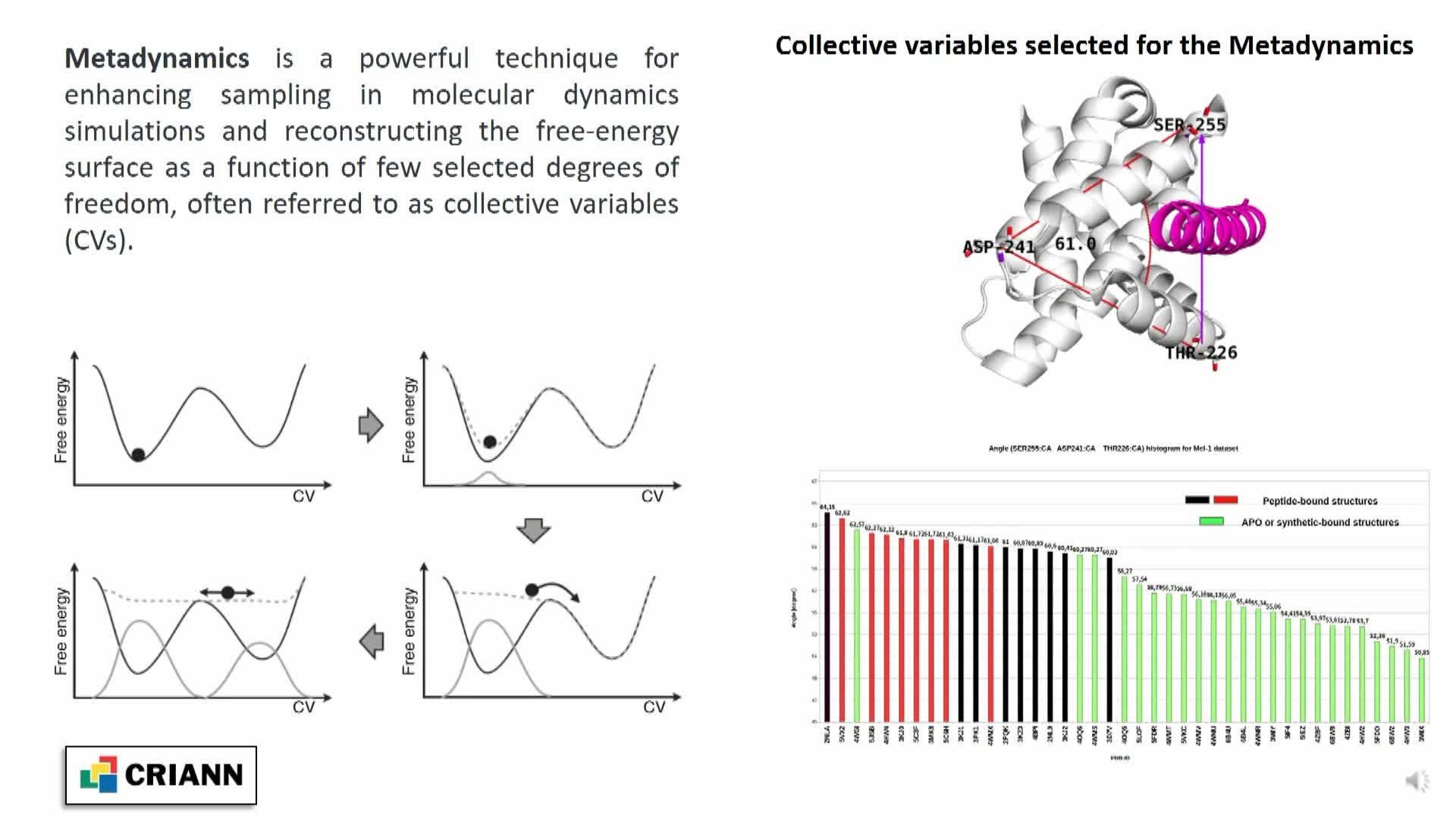
Contexte – VIP (Virtual Imaging Platform) est un portail Web pour l'analyse des données d'imagerie médicale (Glatard et al. 2013). En exploitant les ressources de calcul et de stockage de l'infrastructure EGI, VIP propose aux chercheurs en imagerie médicale l'exécution d'applications scientifiques en tant que services avec la performance des systèmes distribués.
Problématique – La recherche en imagerie médicale traverse actuellement une crise de reproductibilité. En effet, la complexité croissante des procédés d'analyse compromet notre capacité à produire des résultats identiques en réitérant un même traitement sur un même ensemble d'entrées. Au-delà du parti-pris des chercheurs et du processus d'exploration des données (Botvinik-Nezer et al. 2020), l'environnement de calcul (i.e., les librairies statiques, le système d'exploitation, l'infrastructure matérielle) semble jouer un rôle important dans cette crise en ajoutant de l'incertitude numérique aux résultats scientifiques (Glatard et al. 2015). Cette variabilité concerne en particulier la plateforme VIP, qui repose sur des infrastructures de calcul distribué.
Projet – Le projet ANR ReproVIP (ANR-21-CE45-0024-01) aborde cette crise de reproductibilité à tous les niveaux d'analyse, depuis le processus d'exploration des données jusqu'à l'environnement de calcul. Il inclut deux objectifs complémentaires : (i) évaluer l'incertitude des résultats scientifiques obtenus sur la plateforme VIP et (ii) améliorer leur reproductibilité, de manière transparente pour l'utilisateur. Dans cet objectif, des exécutions peuvent être lancées sur des plateformes comme SCIGNE et Grid5000, qui sont susceptibles de fournir un environnement de calcul contrôlé.
Contenu – Cette présentation sera accompagnée d'un poster illustrant les grandes lignes du projet ReproVIP. Nous présenterons ses objectifs opérationnels et les solutions technologiques qui peuvent être mises en place pour améliorer la reproductibilité des analyses en imagerie médicale. Nous introduirons enfin des métriques et une méthodologie d'évaluation de la variabilité des résultats scientifiques au travers des environnements.
Références
• Botvinik-Nezer, Rotem, Felix Holzmeister, Colin F. Camerer, Anna Dreber, Juergen Huber, Magnus Johannesson, Michael Kirchler, et al. 2020. « Variability in the analysis of a single neuroimaging dataset by many teams ». Nature 582 (7810): 84-88. https://doi.org/10.1038/s41586-020-2314-9.
• Glatard, Tristan, Carole Lartizien, Bernard Gibaud, Rafael Ferreira da Silva, Germain Forestier, Frédéric Cervenansky, Martino Alessandrini, et al. 2013. « A Virtual Imaging Platform for Multi-Modality Medical Image Simulation ». IEEE Transactions on Medical Imaging 32 (1): 110-18. https://doi.org/10.1109/TMI.2012.2220154.
• Glatard, Tristan, Lindsay B. Lewis, Rafael Ferreira da Silva, Reza Adalat, Natacha Beck, Claude Lepage, Pierre Rioux, et al. 2015. « Reproducibility of Neuroimaging Analyses across Operating Systems ». Frontiers in Neuroinformatics 9 (avril). https://doi.org/10.3389/fninf.2015.00012.
• Guix-HPC. s. d. « Reproducible computations with Guix ». Dernière consultation le 16 Mai 2022. http://hpc.guix.info/blog/2020/01/reproducible-computations-with-guix/.
Thème
Documentation
Dans la même collection
Sur le même thème
-
Computational reproductibility: an overview illustrated with examples from the medical imaging rese…
PopSorinaComputational reproductibility: an overview illustrated with examples from the medical imaging research community
-
Tutorial Track 1: Reproducible distributed environments with NixOS Compose
Presented by Quentin Guilloteau, Postdoctoral Fellow, Fernando Ayats Llamas, Research Engineer and Olivier Richard, Assistant Professor.
-
Tutorial Track 1: Reproducibility of Scientific Results using E4S Containers
Presented by SHENDE, Sameer, Research Profesor.
-
Tutorial Track2: Fostering Reproducibility By Integrating Large Language Model and Scholarly Knowl…
Presented by Hassan Hussein, PhD Student, Vindoh Ilangovan, Researcher and Kaouter Kebaili, PhD Student.
-
Tutorial Track 3: Managing HPC Software Complexity with Spack
Presented by Massimiliano Culpo, Researcher.
-
Tutorial Track 4: Practical strategies for teaching reproducibility
Presented by Fraida Fund, Research Assistant Professor, Sarah Cohen-Boulakia, Professor and Bogdan Alexandru Stoica, PhD Student.
-
Keynote: Replicable empirical machine learning research
BoulesteixAnne-LaureIn the absence of mathematical theory addressing complex real-life settings beyond simplifying assumptions, the behavior and performance of machine learning methods often has to be addressed by
-
-
-
-
Session 4: Poster Lightning Talks
Talk 1 [00:00] NPF: orchestrate and reproduce network experiments. By Tom Barbette. Presented by Tom Barbette, Assistant Professor. Talk 2 [03:10] : From reproducible to reusable bioinformatics
-
Keynote: Reproducibility and replicability of computer simulations
HinsenKonradSince the early days of the reproducibility crisis, much progress has been made in understanding and improving computational reproducibility and replicability (R and R)...