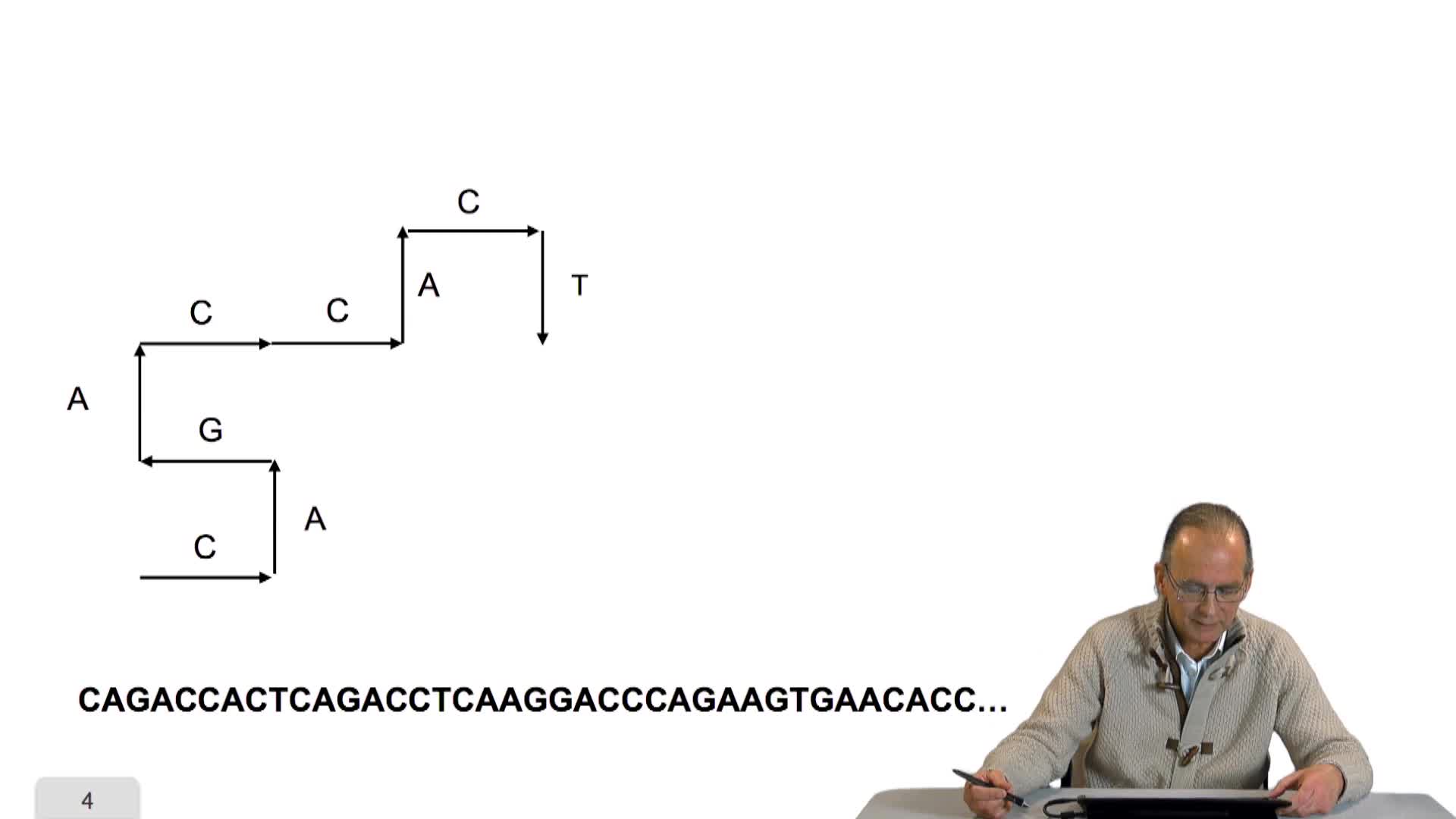Notice
Reasoning over large-scale biological systems with heterogeneous and incomplete data
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
Data produced by the domain of life sciences in the next decade are expected to be highly challenging. In addition to scalability issues which are shared with other applications domains, data produced in life sciences have very specific characteristics: multi-scale, incomplete, heterogeneous but somehow interdependent. This makes data-mining methods less efficient than expected to assist knowledge discovery. An example of such limitations is the study of biological systems in molecular and cellular biology, which cannot be uniquely identified with the data at hand.In this talk, we will introduce a strategy to study biological systems in the framework of incomplete data.
This strategy relies on reasoning and logical programming technics, allowing to model interactions within a system, take into account information carried by the overapproximated dynamics of the system, and finally extract relevant properties by solving combinatorial problems. We will illustrate this approach on the emerging field of systems ecology which aims at understanding interactions between a consortium of microbes and a host organism.
Thème
Documentation
Documents pédagogiques
Sur le même thème
-
Le projet dnarXiv : Stockage de données sur des molécules d'ADN
LavenierDominiqueDuprazElsaLeblancJulienCoatrieuxGouenouDominique Lavenier, Elsa Dupraz, Julien Leblanc et Gouenou Coatrieux nous présentent le projet dnarXiv, un projet porté par le LabEx CominLabs qui explore le stockage de données sur des molécules d
-
21 Molecular Algorithms Using Reprogrammable DNA Self-Assembly
WoodsDamienThe history of computing tells us that computers can be made of almost anything: silicon, gears and levers, neurons, flowing water, interacting particles or even light. Although lithographically
-
Des métiers de la bio-informatique
Courtes vidéos pour sensibiliser le jeune public aux débouchés/métiers de la filière numérique et pour promouvoir les sciences du numérique, plus globalement les carrières scientifiques.L'objectif est
-
Biological Networks Entropies: examples in neural, genetic and social networks
DemongeotJacquesThe networks used in biological applications at different scales (molecular, cellular and populational) are of different types, genetic, neuronal, and social, but they share the same dynamical
-
Génomique et informatique
RislerJean-LoupLa presse généraliste, et bien entendu la presse spécialisée, se font régulièrement l'écho du séquençage complet d'un nouveau génome. Il est cependant impossible pour le grand public de se rendre
-
Apport de l'informatique à la génomique des cancers
ViariAlainLa plupart des gènes de notre génome sont présents en deux copies (une sur chaque chromosome homologue). Dans un génome tumoral, en revanche, il est fréquent d'observer soit des pertes soit, au
-
1.8. Changer l’échelle du chemin
RechenmannFrançoisParmentelatThierryDans la session précédente, je vous ai proposé de m'accompagner dans une balade sur l'ADN. En fait un parcours de la séquence avec un tracé de segments, dont l'orientation dépendait de la lettre
-
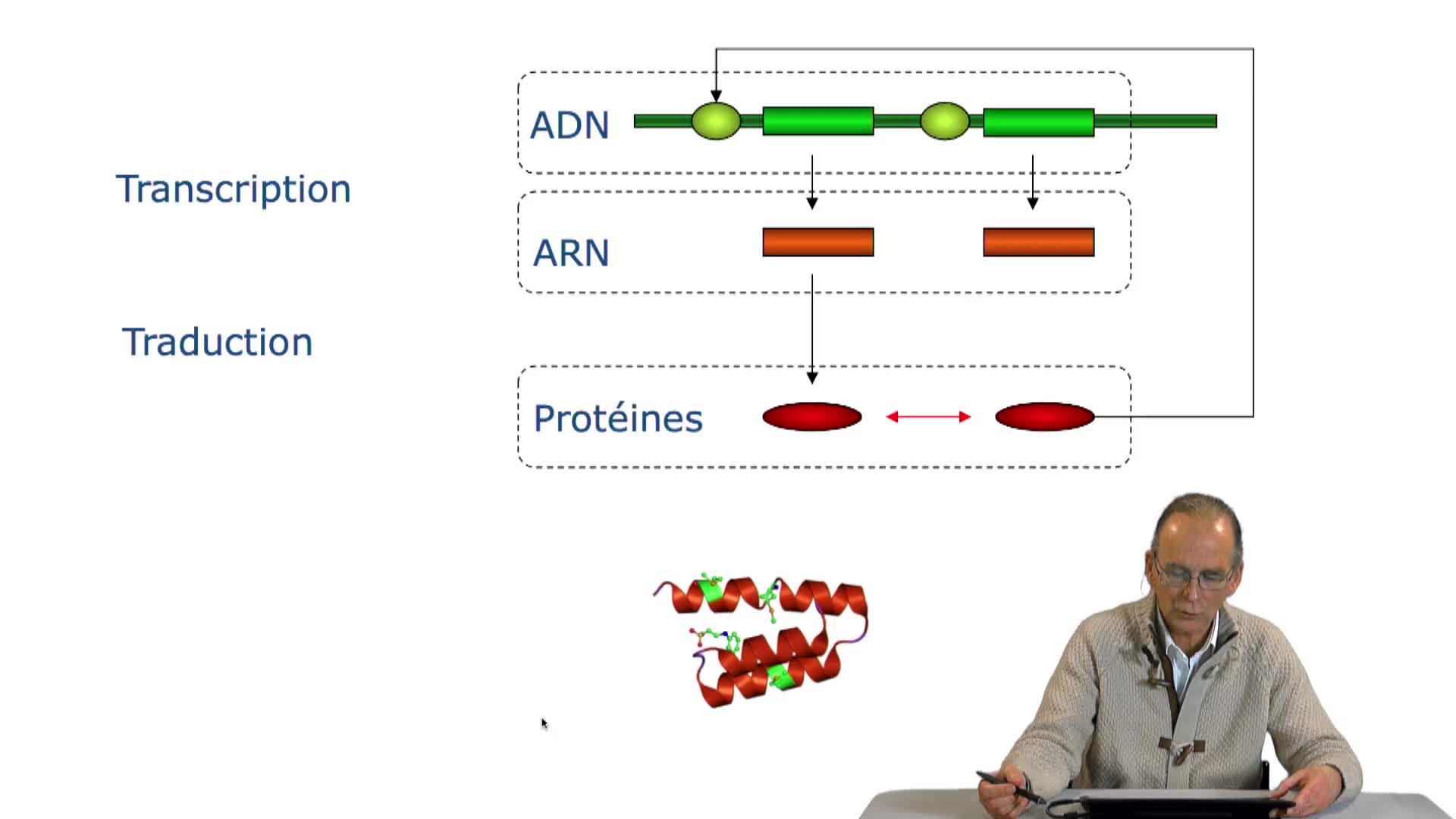
2.3. Le code génétique
RechenmannFrançoisParmentelatThierryGènes et protéines, mais qu'est-ce qu'une protéine ? Une protéine, c'est également une molécule qui est constituée d'une succession de ce que l'on appelle les acides aminés. C'est donc une chaîne d
-
3.6. L’algorithme de Boyer-Moore
RechenmannFrançoisParmentelatThierryVous avez compris que la recherche de motifs, c'est-à-dire de sous-chaînes de caractères dans une chaîne plus importante, était un composant important de beaucoup d'algorithmes de bio-informatique.
-
4.1. Comment prédire les fonctions des gènes/protéines ?
RechenmannFrançoisParmentelatThierryAprès avoir regardé dans les yeux, les semaines précédentes, l'ADN, vu comment cet ADN par séquençage produisait des textes, des séquences génomiques, étudié la relation entre gènes et protéines,
-
5.4. L’algorithme UPGMA
RechenmannFrançoisParmentelatThierryL'algorithme, que nous allons étudier pour la reconstruction d'arbres phylogénétiques à partir des distances, s'appelle UPGMA. Un nom plutôt compliqué pour une méthode qui est plutôt simple. Et même,
-
5.6. La diversité des algorithmes informatiques
RechenmannFrançoisParmentelatThierryNous n'avons vu dans ce cours qu'un exemple extrêmement réduit d'algorithme bio informatique. Il existe en effet une très grande diversité de ces algorithmes bio informatiques qui sont motivés par l