Notice
4.7. Alignment costs
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
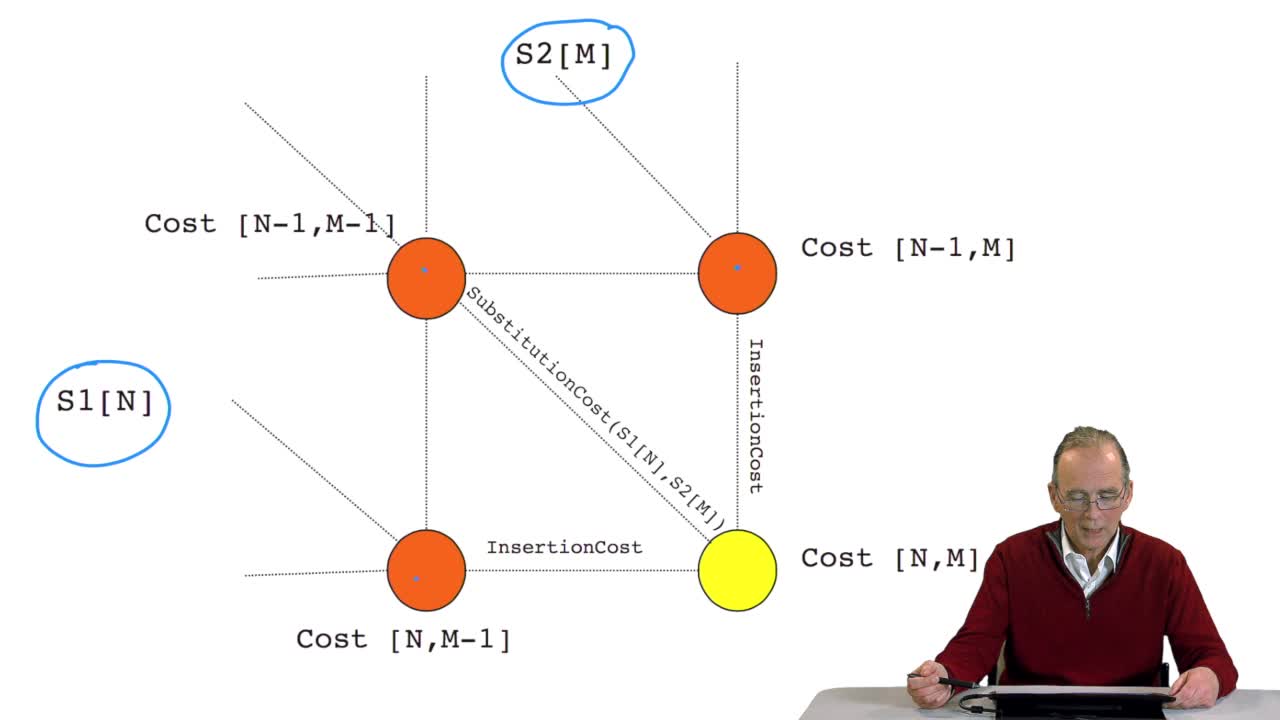
We have seen how we can compute the cost of the path ending on the last node of our grid if we know the cost of the sub-path ending on the three adjacent nodes. It is time now to see more deeply why these costs are used to compute the cost in the last node. So again, we saw how we can compute the cost here of the path ending on that node if we know the cost of the sub-path ending on these three red nodes. Indeed, if we come from that node, the cost on that node will be the cost of that node plus the cost of the gap that is an insertion cost. If we come from that node, the situation is similar, the cost of that node will be the cost of thatone plus the same insertion cost. If we come from that node, thecost here will be the sum of the cost of that node plus the code of the substitution of the letter N of the sequence by letter M of the second sequence. And this cost is known withinthe substitution matrix, here, which tells for each pair of characters, here in the four letter alphabet, the cost of substituting oneletter, one nucleotide by another one. So, now since we want to havehere the minimal cost what we do is we take, we return as the value of the cost here the minimum of these three expressions.
Thème
Documentation
Dans la même collection
-
4.6. A path is optimal if all its sub-paths are optimal
RECHENMANN François
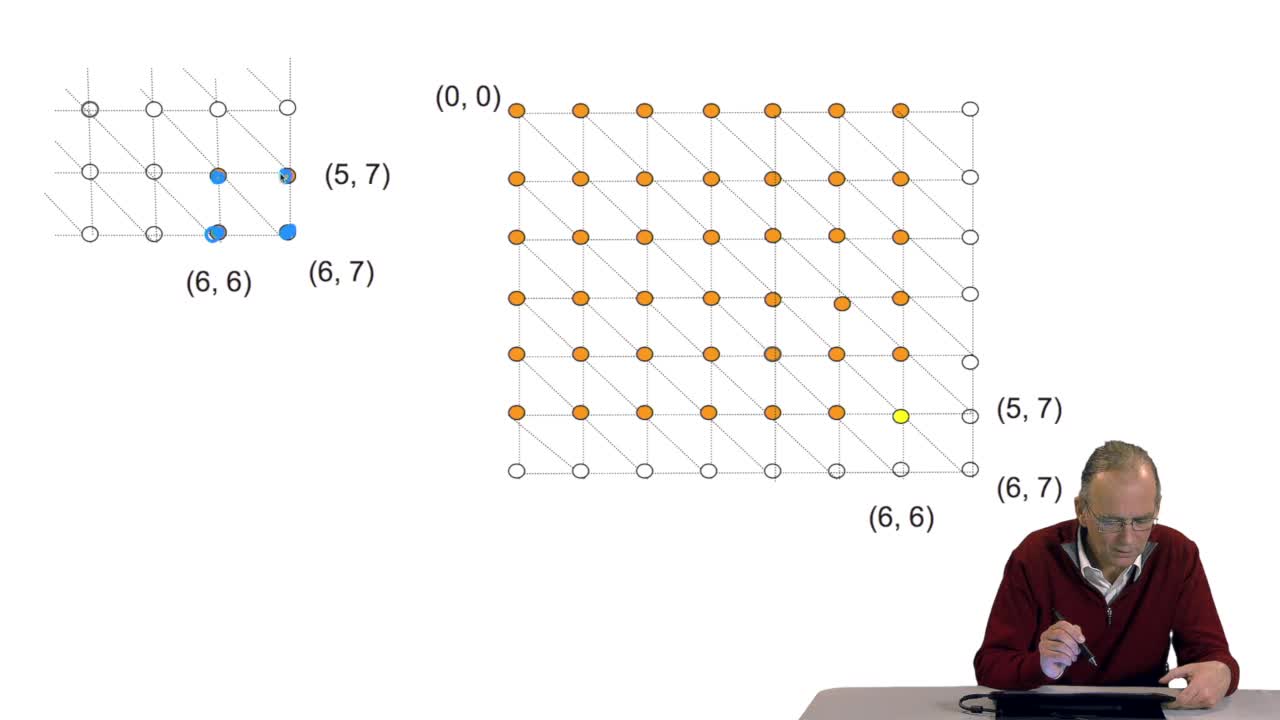
A sequence alignment between two sequences is a path in a grid. So that, an optimal sequence alignmentis an optimal path in the same grid. We'll see now that a property of this optimal path provides
-
4.1. How to predict gene/protein functions?
RECHENMANN François
Last week we have seen that annotating a genome means first locating the genes on the DNA sequences that is the genes, the region coding for proteins. But this is indeed the first step,the next very
-
4.10. How efficient is this algorithm?
RECHENMANN François
We have seen the principle of an iterative algorithm in two paths for aligning and comparing two sequences of characters, here DNA sequences. And we understoodwhy the iterative version is much more
-
4.4. Aligning sequences is an optimization problem
RECHENMANN François
We have seen a nice and a quitesimple solution for measuring the similarity between two sequences. It relied on the so-called hammingdistance that is counting the number of differencesbetween two
-
4.8. A recursive algorithm
RECHENMANN François
We have seen how we can computethe optimal cost, the ending node of our grid if we know the optimal cost of the three adjacent nodes. This is this computation scheme we can see here using the notation
-
4.2. Why gene/protein sequences may be similar?
RECHENMANN François
Before measuring the similaritybetween the sequences, it's interesting to answer the question: why gene or protein sequences may be similar? It is indeed veryinteresting because the answer is related
-
4.5. A sequence alignment as a path
RECHENMANN François
Comparing two sequences and thenmeasuring their similarities is an optimization problem. Why? Because we have seen thatwe have to take into account substitution and deletion. During the alignment, the
-
4.9. Recursion can be avoided: an iterative version
RECHENMANN François
We have written a recursive function to compute the optimal path that is an optimal alignment between two sequences. Here all the examples I gave were onDNA sequences, four letter alphabet. OK. The
-
4.3. Measuring sequence similarity
RECHENMANN François
So we understand why gene orprotein sequences may be similar. It's because they evolve togetherwith the species and they evolve in time, there aremodifications in the sequence and that the sequence
Avec les mêmes intervenants et intervenantes
-
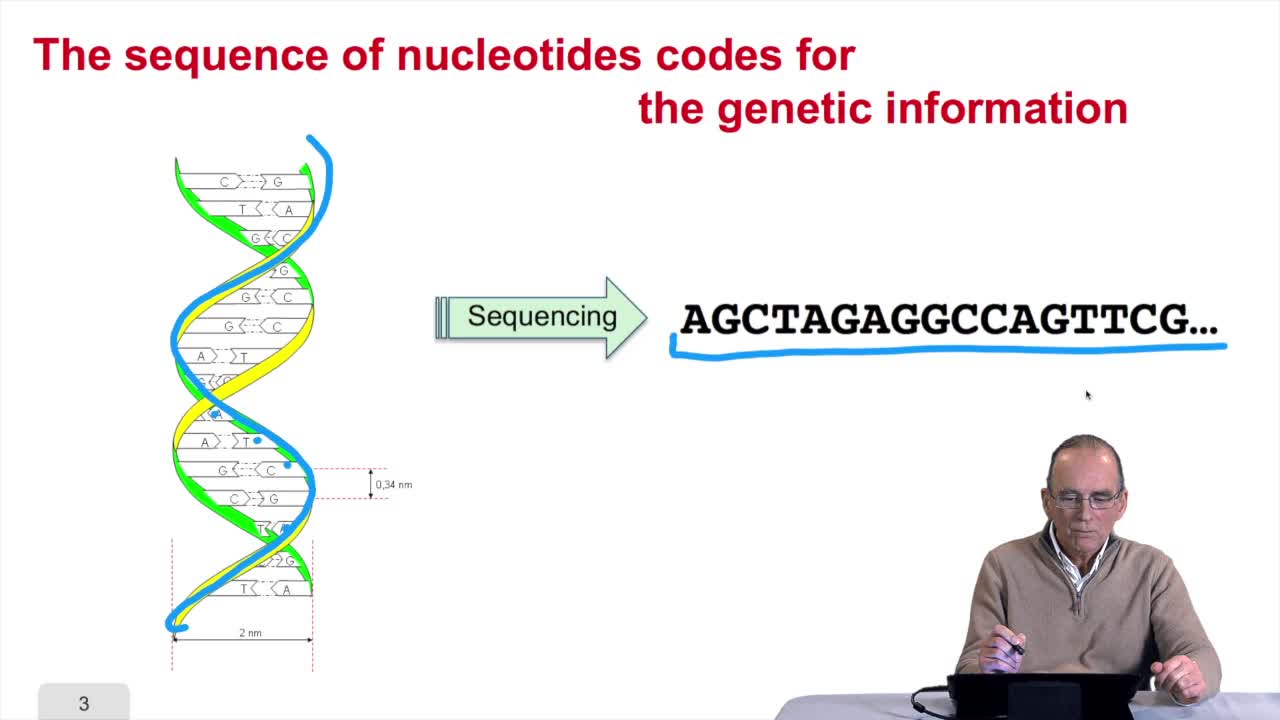
1.3. DNA codes for genetic information
RECHENMANN François
Remember at the heart of any cell,there is this very long molecule which is called a macromolecule for this reason, which is the DNA molecule. Now we will see that DNA molecules support what is called
-
2.1. The sequence as a model of DNA
RECHENMANN François
Welcome back to our course on genomes and algorithms that is a computer analysis ofgenetic information. Last week we introduced the very basic concept in biology that is cell, DNA, genome, genes
-
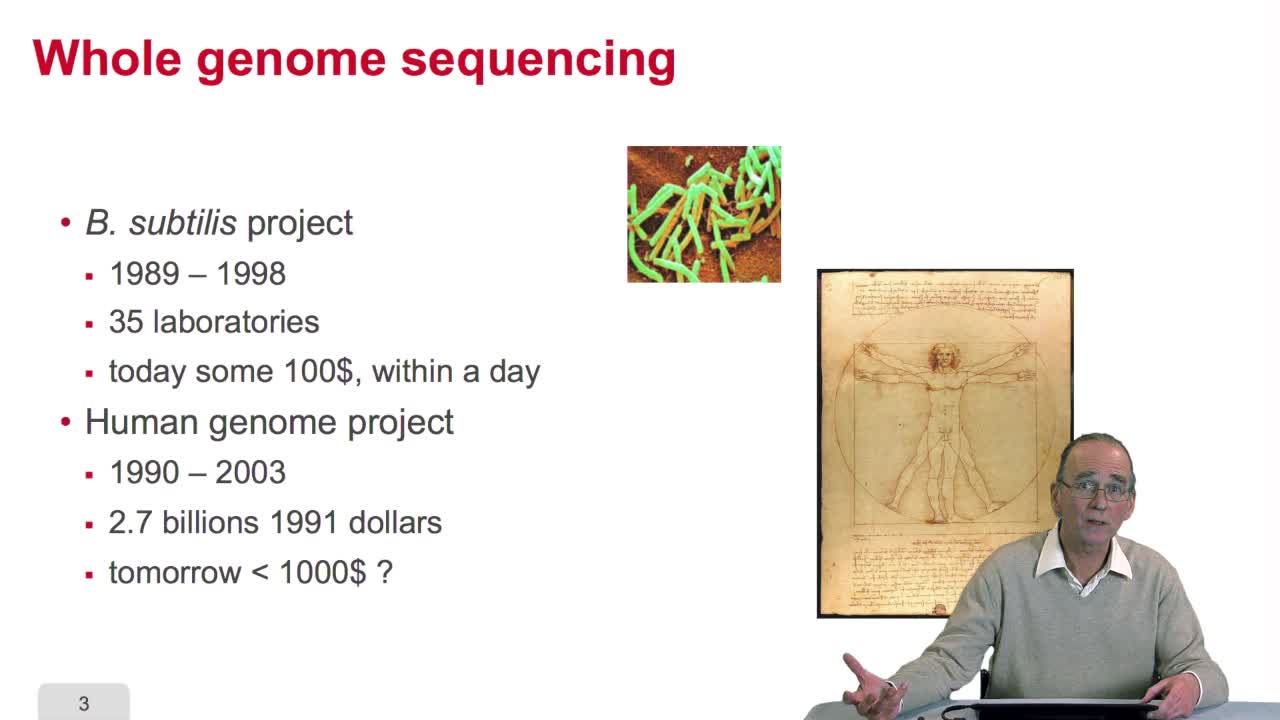
2.9. Whole genome sequencing
RECHENMANN François
Sequencing is anexponential technology. The progresses in this technologyallow now to a sequence whole genome, complete genome. What does it mean? Well let'stake two examples: some twenty years ago,
-
3.7. Index and suffix trees
RECHENMANN François
We have seen with the Boyer-Moore algorithm how we can increase the efficiency of spin searching through the pre-processing of the pattern to be searched. Now we will see that an alternative way of
-
4.3. Measuring sequence similarity
RECHENMANN François
So we understand why gene orprotein sequences may be similar. It's because they evolve togetherwith the species and they evolve in time, there aremodifications in the sequence and that the sequence
-
5.3. Building an array of distances
RECHENMANN François
So using the sequences of homologous gene between several species, our aim is to reconstruct phylogenetic tree of the corresponding species. For this, we have to comparesequences and compute distances
-
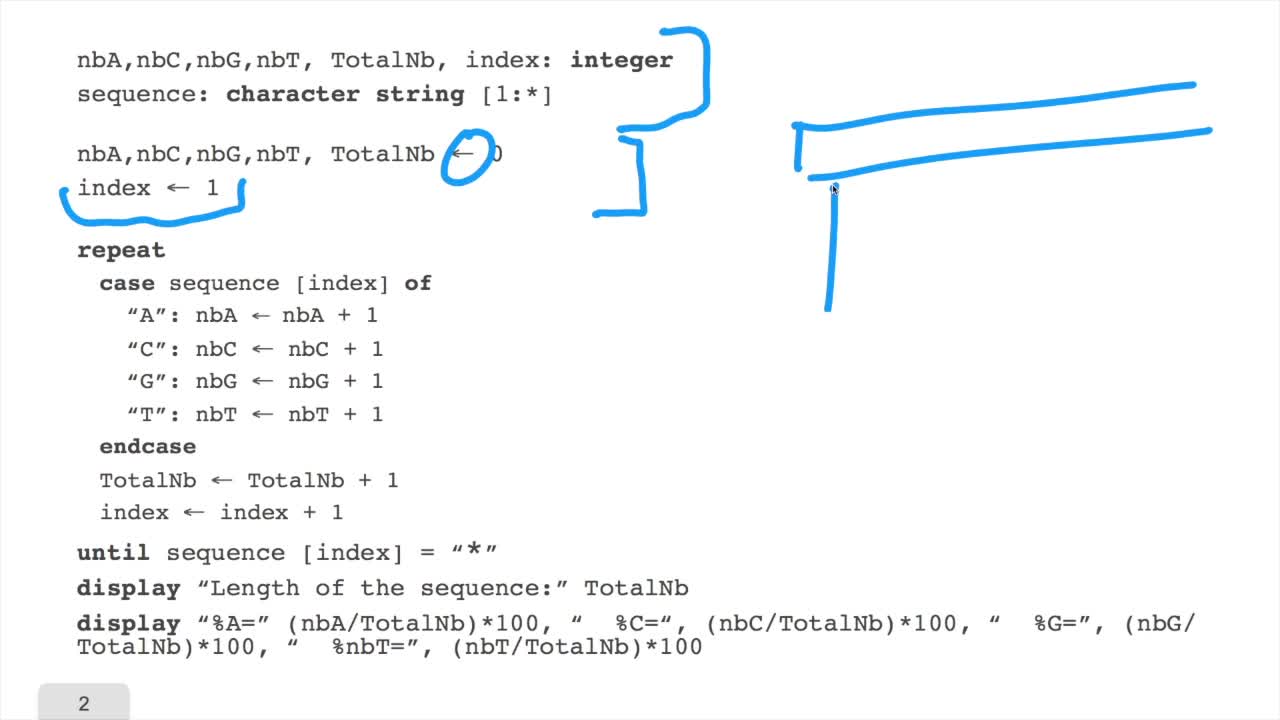
1.6. GC and AT contents of DNA sequence
RECHENMANN François
We have designed our first algorithmfor counting nucleotides. Remember, what we have writtenin pseudo code is first declaration of variables. We have several integer variables that are variables which
-
2.5. Implementing the genetic code
RECHENMANN François
Remember we were designing our translation algorithm and since we are a bit lazy, we decided to make the hypothesis that there was the adequate function forimplementing the genetic code. It's now time
-
3.2. A simple algorithm for gene prediction
RECHENMANN François
Based on the principle we statedin the last session, we will now write in pseudo code a firstalgorithm for locating genes on a bacterial genome. Remember first how this algorithm should work, we first
-
3.10. Gene prediction in eukaryotic genomes
RECHENMANN François
If it is possible to have verygood predictions for bacterial genes, it's certainly not the caseyet for eukaryotic genomes. Eukaryotic cells have manydifferences in comparison to prokaryotic cells. You
-
4.10. How efficient is this algorithm?
RECHENMANN François
We have seen the principle of an iterative algorithm in two paths for aligning and comparing two sequences of characters, here DNA sequences. And we understoodwhy the iterative version is much more
-
5.7. The application domains in microbiology
RECHENMANN François
Bioinformatics relies on many domains of mathematics and computer science. Of course, algorithms themselves on character strings are important in bioinformatics, we have seen them. Algorithms and