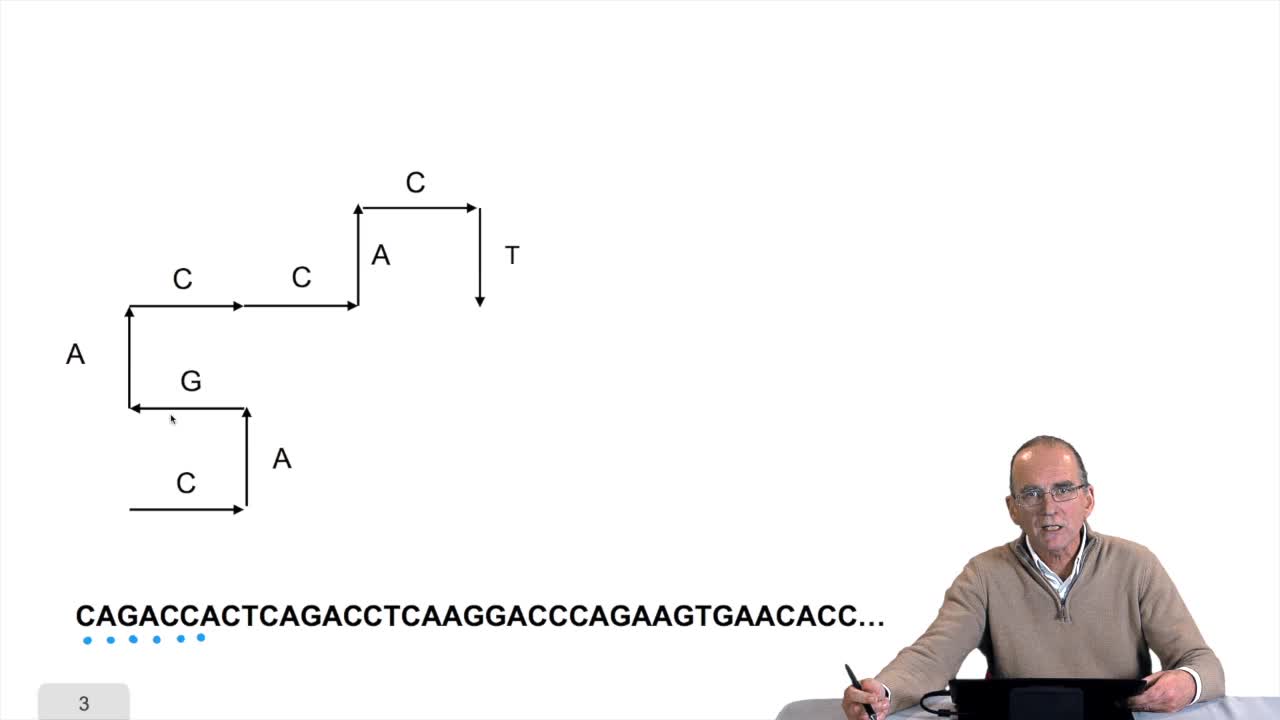Notice
2.7. Les compromis de la conception d’algorithmes
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
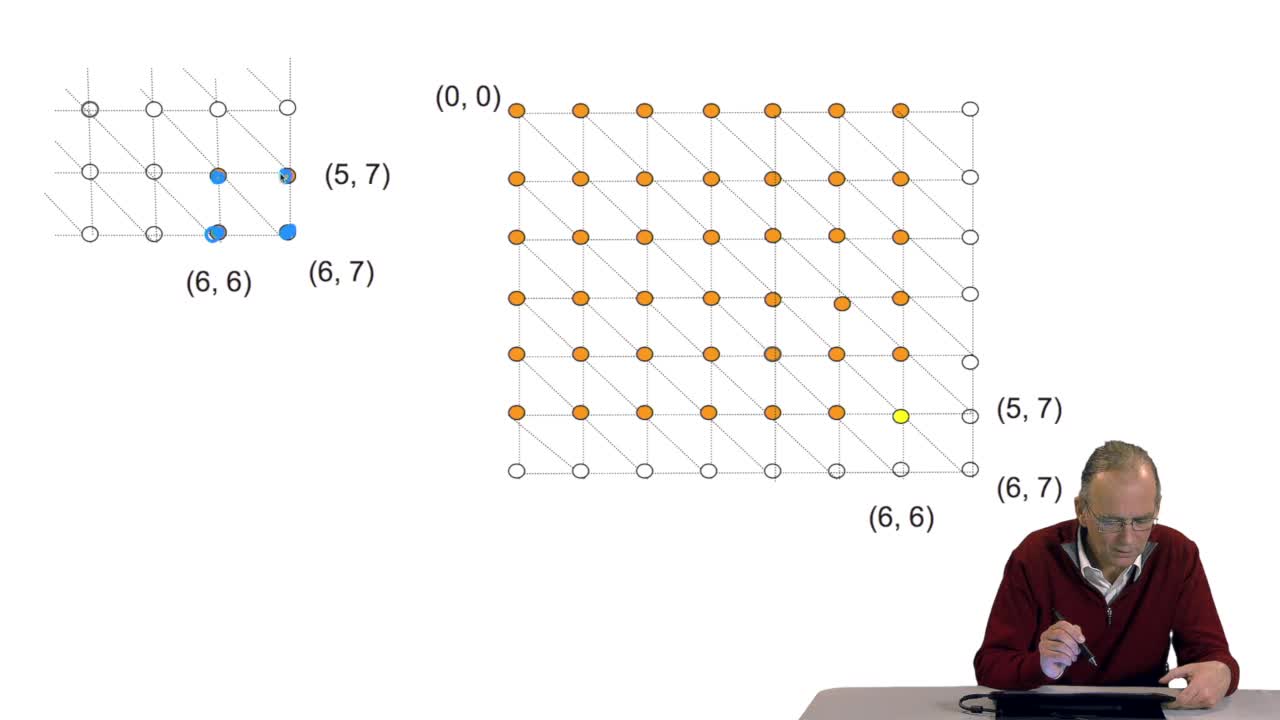
La mise en oeuvre d'une structure de données appropriée permet, nous l'avons vu, d'améliorer les performances d'algorithmes. Nous en avons vu l'exemple sur la recherche d'un triplet dans un tableau de code génétique, quand nous avons ajouté ces tables d'index, nous avons vu que nous avons diminué de façon tout à fait significative, le nombre de comparaisons à effectuer. Je vous propose maintenant une autre approche où les index ne sont pas sous forme de table mais sont calculés. Il faut que vous vous souveniez de la manière dont le tableau est organisé. D'abord tous les triplets qui commencent par T, tous ceux qui commencent par C, par A, par G. À l'intérieur de cette partie-là du tableau, d'abord tous les triplets dont la deuxième lettre est un T, et cetera, et cetera. Nous allons tirer partie encore une fois de cette organisation, mais d'une manière différente de ce que nous avons fait jusqu'à présent...
Intervention / Responsable scientifique
Thème
Documentation
Dans la même collection
-
2.8. Les technologies de séquençage de l’ADN
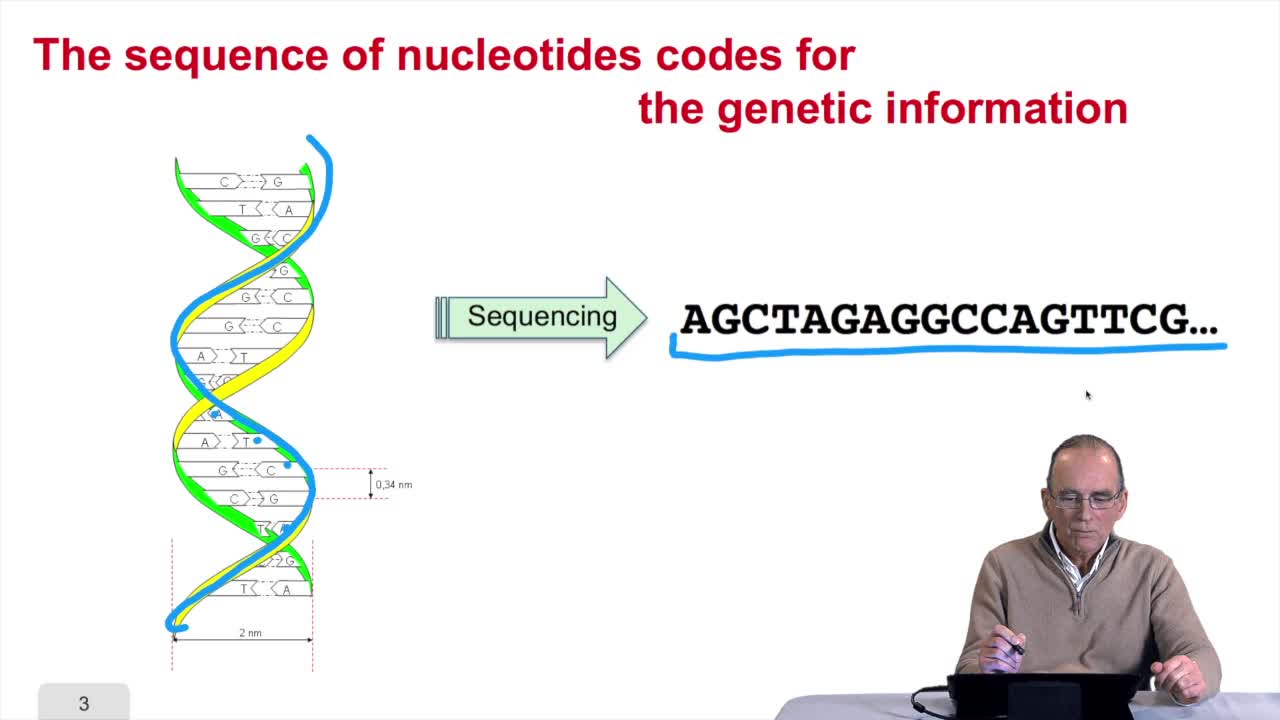
RechenmannFrançoisParmentelatThierryNous parlons beaucoup dans ce cours de séquences génomiques ou séquences d'ADN, que nous voyons pour des raisons algorithmiques sous forme de chaînes de caractères. Comment ces séquences, ces chaînes
-
2.2. Les gènes, de Mendel à la biologie moléculaire
RechenmannFrançoisParmentelatThierryLa séquence de caractères est un bon modèle de l'ADN, un des modèles possibles de l'ADN et il est bon parce qu'il est utile. On va voir en particulier que ce modèle simple peut servir de support à de
-
2.5. Implémenter le code génétique
RechenmannFrançoisParmentelatThierryNous avons écrit le corps de l'algorithme de traduction, et nous avons fractionné la complexité d'écriture de cet algorithme en faisant appel à une fonction qui recherche dans le tableau, qui
-
2.9. Le séquençage de génomes complets
RechenmannFrançoisParmentelatThierryLes progrès dans les technologies de séquençage ont permis d'aborder le séquençage complet de génome. Là encore, les progrès ont été spectaculaires. Prenons l'exemple du projet de séquençage de la
-
2.3. Le code génétique
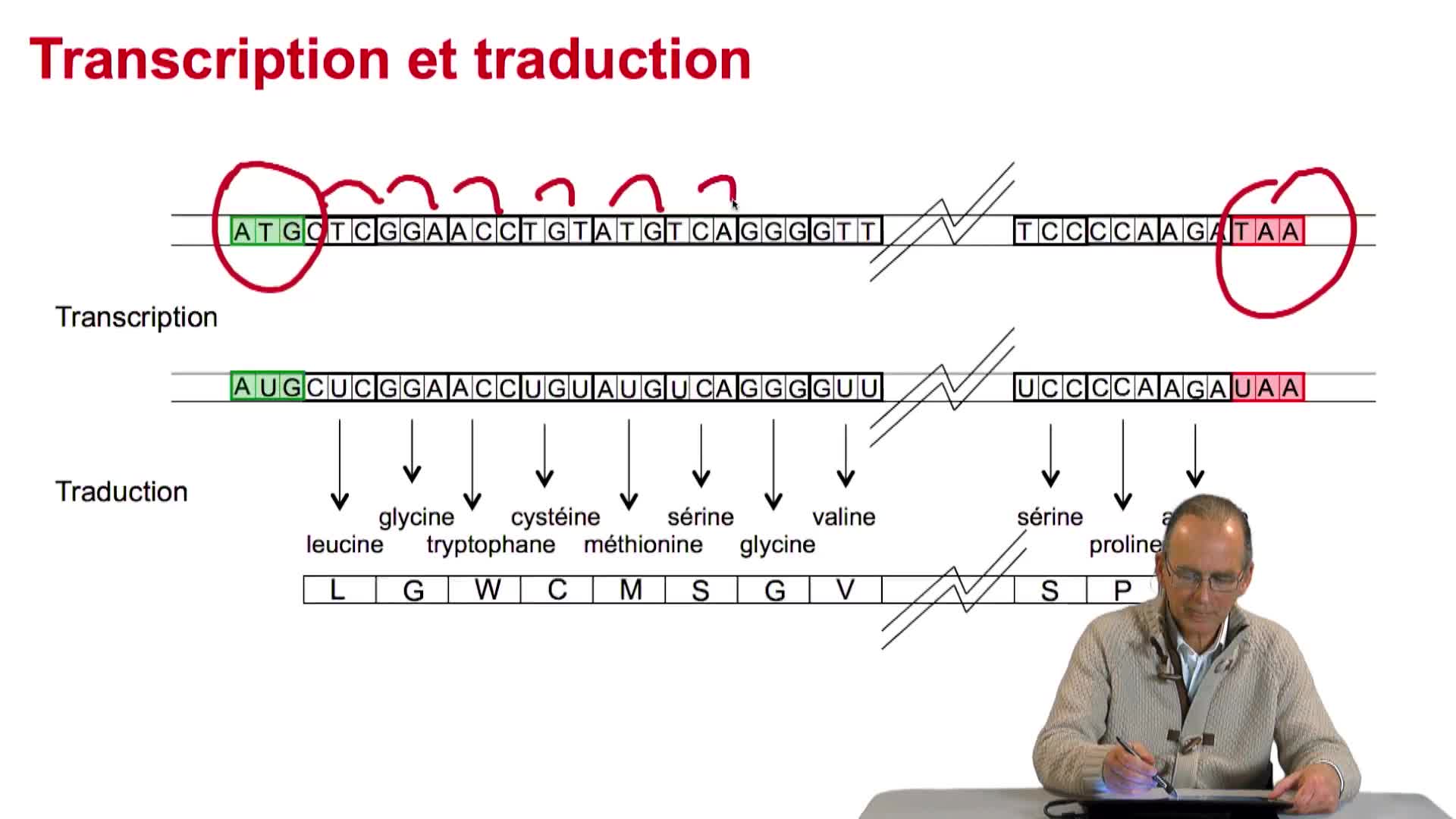
RechenmannFrançoisParmentelatThierryGènes et protéines, mais qu'est-ce qu'une protéine ? Une protéine, c'est également une molécule qui est constituée d'une succession de ce que l'on appelle les acides aminés. C'est donc une chaîne d
-
2.6. Algorithmes + structures de données = programmes
RechenmannFrançoisParmentelatThierryEn écrivant le code de la fonction, qui recherche un triplet dans le tableau qui implémente le code génétique, nous avons terminé et obtenu un algorithme de traduction d'une séquence d'ADN, voire d
-
2.1. La séquence est-elle un bon modèle de l’ADN ?
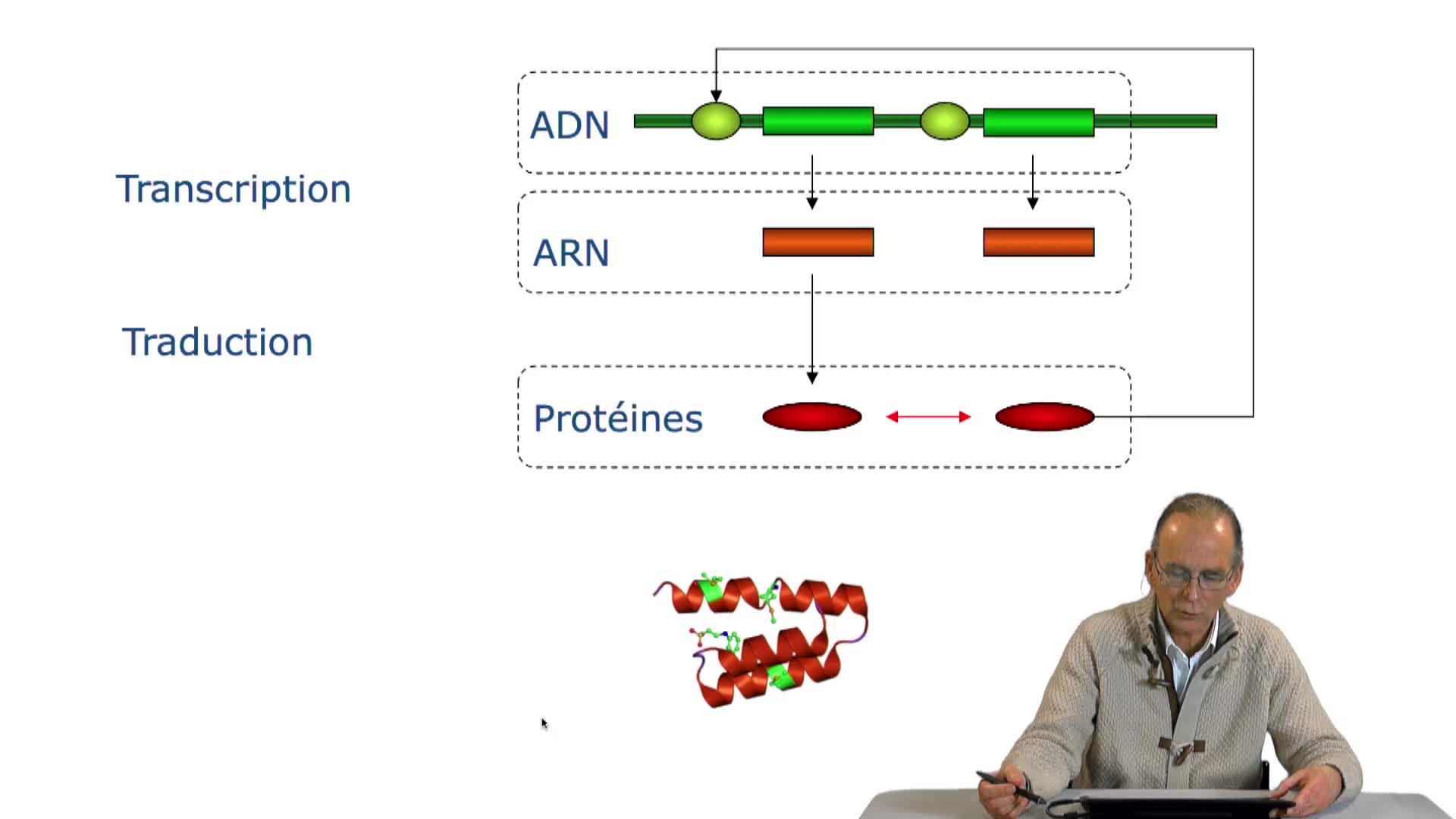
RechenmannFrançoisParmentelatThierryL'ADN porte l'information génétique, plus précisément l'ADN porte les gènes, c'est-à-dire les régions de cette molécule qui portent l'information utilisée par la cellule pour synthétiser les protéines
-
2.10. Comment trouver les gènes ?
RechenmannFrançoisParmentelatThierryL'obtention de la séquence complète d'un génome d'un organisme vivant est certes un beau résultat, mais c'est en fait le début d'une longue phase d'interprétation, d'annotations et de comparaisons.
-
2.4. Un algorithme de traduction
RechenmannFrançoisParmentelatThierryUne protéine, en tant que succession d'acides aminés, peut-être vue comme le résultat d'un processus de traduction d'une chaîne de caractères écrite dans un alphabet de 4 lettres en une autre chaîne
Avec les mêmes intervenants et intervenantes
-
1.5. Counting nucleotides
RechenmannFrançoisIn this session, don't panic. We will design our first algorithm. This algorithm is forcounting nucleotides. The idea here is that as an input,you have a sequence of nucleotides, of bases, of letters,
-
2.4. A translation algorithm
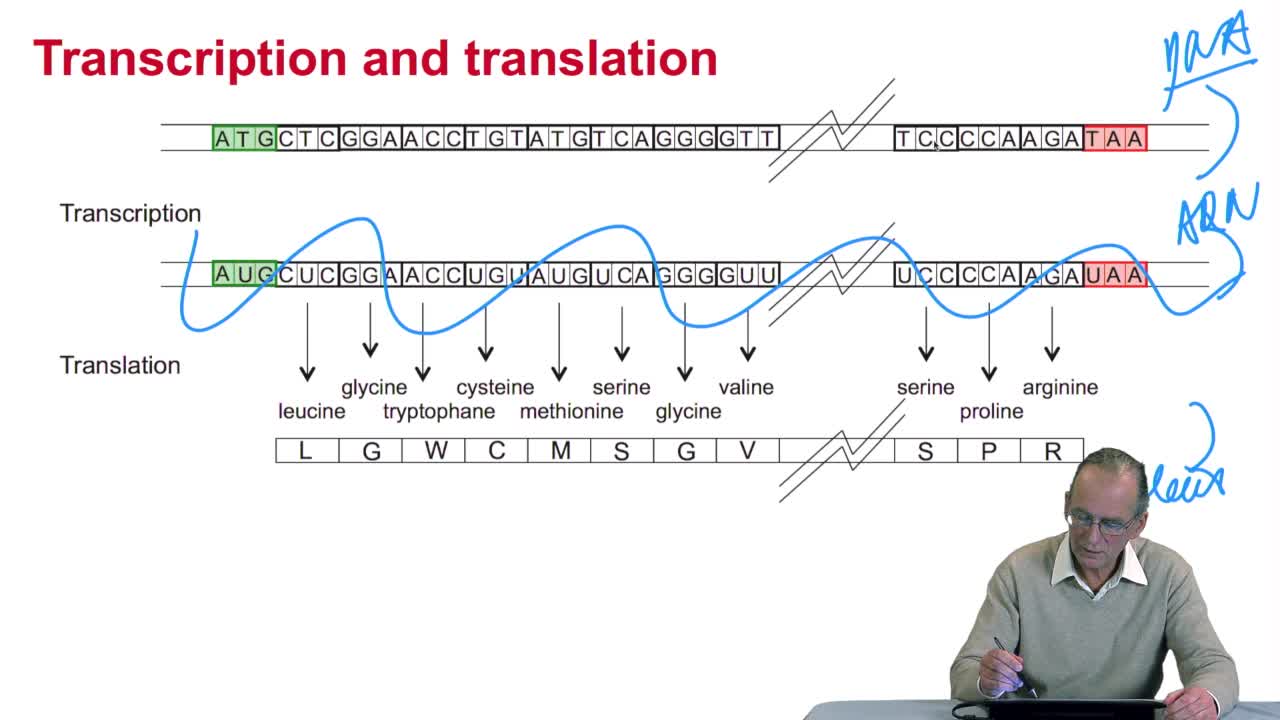
RechenmannFrançoisWe have seen that the genetic codeis a correspondence between the DNA or RNA sequences and aminoacid sequences that is proteins. Our aim here is to design atranslation algorithm, we make the
-
3.1. All genes end on a stop codon
RechenmannFrançoisLast week we studied genes and proteins and so how genes, portions of DNA, are translated into proteins. We also saw the very fast evolutionof the sequencing technology which allows for producing
-
3.9. Benchmarking the prediction methods
RechenmannFrançoisIt is necessary to underline that gene predictors produce predictions. Predictions mean that you have no guarantees that the coding sequences, the coding regions,the genes you get when applying your
-
4.2. Why gene/protein sequences may be similar?
RechenmannFrançoisBefore measuring the similaritybetween the sequences, it's interesting to answer the question: why gene or protein sequences may be similar? It is indeed veryinteresting because the answer is related
-
5.4. The UPGMA algorithm
RechenmannFrançoisWe know how to fill an array with the values of the distances between sequences, pairs of sequences which are available in the file. This array of distances will be the input of our algorithm for
-
1.8. Compressing the DNA walk
RechenmannFrançoisWe have written the algorithm for the circle DNA walk. Just a precision here: the kind of drawing we get has nothing to do with the physical drawing of the DNA molecule. It is a symbolic
-
2.7. The algorithm design trade-off
RechenmannFrançoisWe saw how to increase the efficiencyof our algorithm through the introduction of a data structure. Now let's see if we can do even better. We had a table of index and weexplain how the use of these
-
3.4. Predicting all the genes in a sequence
RechenmannFrançoisWe have written an algorithm whichis able to locate potential genes on a sequence but only on one phase because we are looking triplets after triplets. Now remember that the genes maybe located on
-
4.7. Alignment costs
RechenmannFrançoisWe have seen how we can compute the cost of the path ending on the last node of our grid if we know the cost of the sub-path ending on the three adjacent nodes. It is time now to see more deeply why
-
4.9. Recursion can be avoided: an iterative version
RechenmannFrançoisWe have written a recursive function to compute the optimal path that is an optimal alignment between two sequences. Here all the examples I gave were onDNA sequences, four letter alphabet. OK. The
-
1.3. DNA codes for genetic information
RechenmannFrançoisRemember at the heart of any cell,there is this very long molecule which is called a macromolecule for this reason, which is the DNA molecule. Now we will see that DNA molecules support what is called
Sur le même thème
-
La voix, une donnée identifiante à protéger
VincentEmmanuelEmmanuel Vincent, chercheur au Centre Inria de l'Université de Lorraine et au Loria (Laboratoire lorrain de recherche en informatique et ses applications), présente sa recherche sur l'anonymisation de
-
Podcast 1/4 d'heure avec : Emmanuel Vincent, chercheur au Centre Inria de l'Université de Lorraine …
VincentEmmanuelRencontre avec Emmanuel Vincent - chercheur au Centre Inria de l'Université de Lorraine et Loria (Laboratoire lorrain de recherche en informatique et ses applications).
-
Stockage de données numériques sur ADN synthétique : Introduction au domaine
AntoniniMarcDuprazElsaLavenierDominiquePrésentation globale des différentes étapes du stockage de données sur des molécules d'ADN synthétique
-
Stockage de données numériques sur ADN synthétique : Production des données: synthèse, séquençage
LavenierDominiqueBarbryPascalDescription des opérations d'écriture et de lecture des molécules d'ADN : synthèse et séquençage.
-
Stockage de données numériques sur ADN synthétique : Reconstruction des données
LavenierDominiqueTraitement des données après séquençage
-
Stockage de données numériques sur ADN synthétique : Codage Canal
DuprazElsaTechniques de codage pour le stockage de données sur ADN
-
Stockage de données numériques sur ADN synthétique : Codage Source
AntoniniMarcCodage source pour le stockage de données sur ADN synthétique
-
Stockage de données numériques sur ADN synthétique : Théorie de l'information
Kas HannaSergeQuelle quantité d'information peut-on stocker et récupérer de manière fiable dans l'ADN ?
-
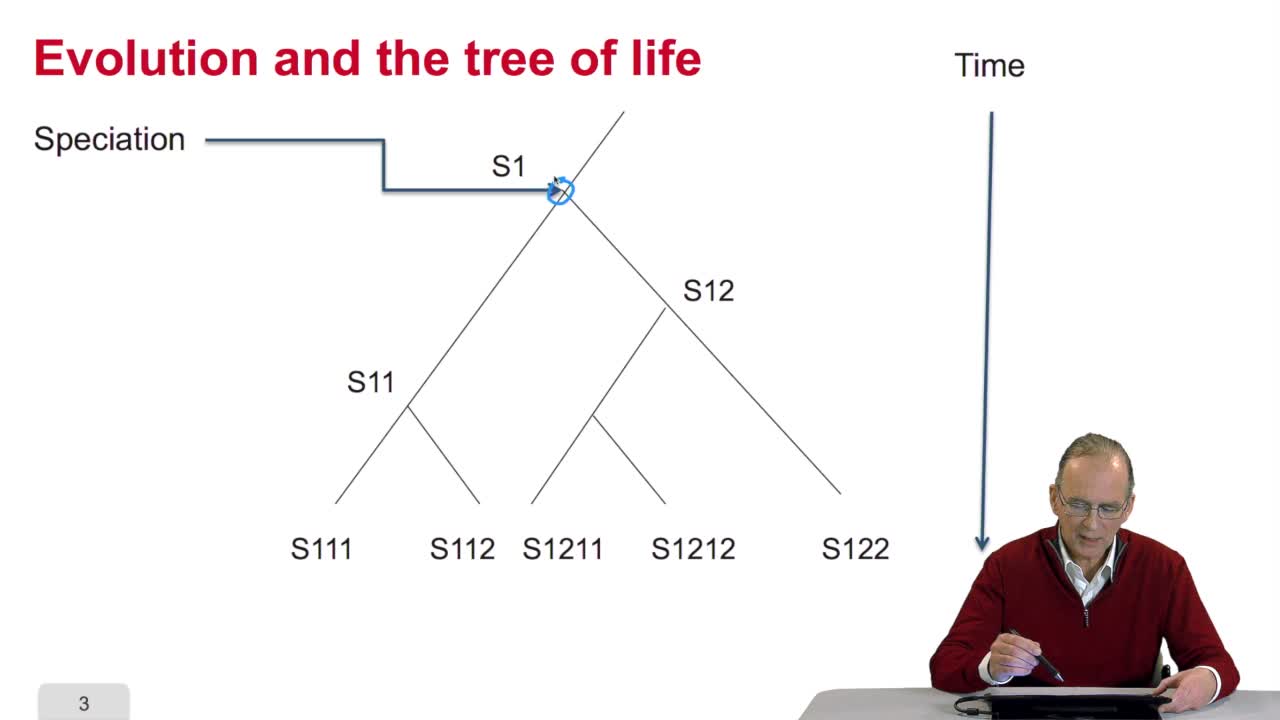
The tree of life
AbbySophieLes Rencontres Exobiologiques pour Doctorants (RED) sont une école de formation sur les « bases de l'astrobiologie ». L’édition 2025 s’est tenue du 16 au 21 mars au Parc Ornithologique du Teich.
-
Machines algorithmiques, mythes et réalités
MazenodVincentVincent Mazenod, informaticien, partage le fruit de ses réflexions sur l'évolution des outils numériques, en lien avec les problématiques de souveraineté, de sécurité et de vie privée...
-
Désassemblons le numérique - #Episode11 : Les algorithmes façonnent-ils notre société ?
SchwartzArnaudLima PillaLaércioEstériePierreSalletFrédéricFerbosAudeRoumanosRayyaChraibi KadoudIkramUn an après le tout premier hackathon sur les méthodologies d'enquêtes journalistiques sur les algorithmes, ce nouvel épisode part à la rencontre de différents points de vue sur les algorithmes.
-
Les machines à enseigner. Du livre à l'IA...
BruillardÉricQue peut-on, que doit-on déléguer à des machines ? C'est l'une des questions explorées par Éric Bruillard qui, du livre aux IA génératives, expose l'évolution des machines à enseigner...