Notice
3.8. Des méthodes probabilistes à la rescousse
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
Nous avons vu comment la qualité des prédictions de gènes dans un génome bactérien, pouvait être améliorée à travers la recherche d'occurrences de motifs particuliers liés au site de fixation du ribosome, le fameux site RBS.
Il n'en reste pas moins que ces prédictions de gènes, basée uniquement sur la recherche de motifs, sont insuffisantes pour produire des prédictions de qualité satisfaisante. Les bio informaticiens complètent donc cette approche-là par des approches probabilistes qui permettent de tester le caractère codant, ou non codant, de la région prédite. Voyons de plus près comment ces méthodes fonctionnent.
Pour ce faire je vais prendre un exemple qui n'a rien à voir avec la génomique. Un exemple tout à fait hypothétique mais intéressant dans son principe. Supposons donc que vous disposiez d'un texte. écrit à l'aide des 26 lettres de notre alphabet habituel, et la majorité de ce texte est écrit de façon aléatoire. Les lettres apparaissent au hasard, sauf sur certains passages qui sont écrits dans un langage, c'est-à-dire une langue humaine, parlée. Est-il possible de détecter automatiquement ces passages ? La réponse est oui en utilisant le fait que la répartition des lettres, la fréquence des lettres dans un texte écrit dans une langue naturelle, telle que le français ou l'anglais n'est évidemment pas aléatoire. Qui plus est on connaît par des études statistiques, les probabilités d'occurrence, par exemple de la lettre E en français ou la lettre W en français comme en anglais...
Intervention / Responsable scientifique
Dans la même collection
-
3.4. Prédiction de tous les gènes d’une séquence
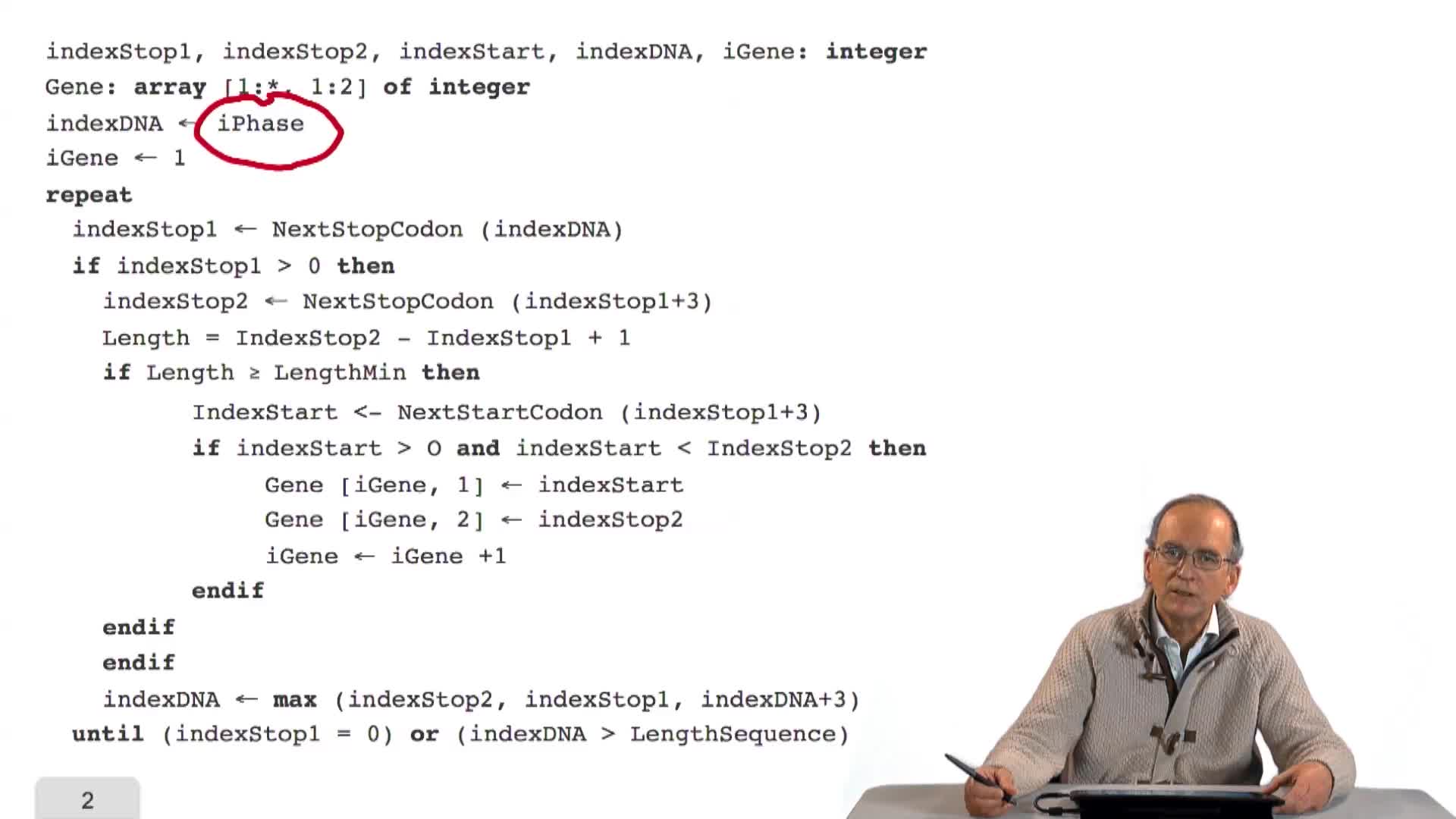
RechenmannFrançoisParmentelatThierryEn combinant de façon adéquate la recherche des triplés Stop et Start sur un brin d'ADN, nous avons obtenu un algorithme qui prédit les gènes sur ce brin, mais également sur une phase. C'est-à-dire en
-
3.7. Index et arbre des suffixes
RechenmannFrançoisParmentelatThierryIl y a donc deux approches pour améliorer la performance des algorithmes de recherche d'un motif dans une chaîne de caractères. La première approche consiste à pré-traiter le motif. On a vu un exemple
-
3.2. Un algorithme simple de prédiction de gènes
RechenmannFrançoisParmentelatThierrySur la base des principes énoncés précédemment, nous allons écrire un premier algorithme de prédiction de gènes sur un texte génomique procaryote. Je rappelle ces principes. L'idée est la suivante :
-
3.5. Comment améliorer la qualité des prédictions ?
RechenmannFrançoisParmentelatThierryIl faut toujours le répéter et le souligner, les algorithmes qui déterminent des gènes déterminent des gènes candidats. Ce sont des prédictions de gènes. Donc la question est de savoir s'il est
-
3.9. Comment évaluer la qualité de prédiction des méthodes ?
RechenmannFrançoisParmentelatThierryNous avons vu qu'il était possible, ou du moins nous le pensions, améliorer la qualité de prédiction des gènes sur un génome bactérien en introduisant des démarches supplémentaires, de recherches de
-
3.3. À la recherche des codons start et stop
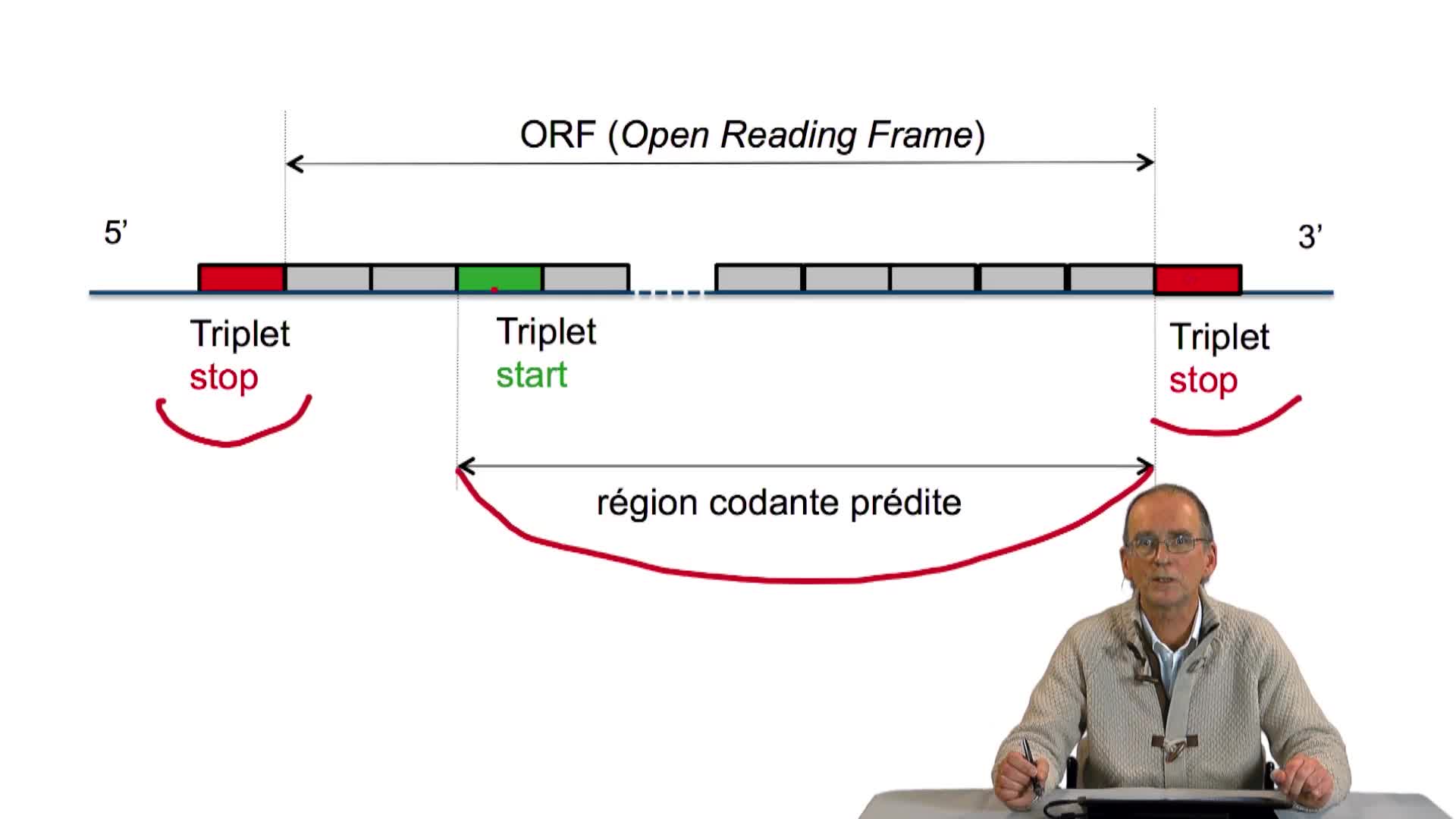
RechenmannFrançoisParmentelatThierryNous avons écrit la structure, l'ossature d'un algorithme de prédiction de gènes dans un génome bactérien, en utilisant les principes que nous avions énoncés précédemment. Cet algorithme est incomplet
-
3.6. L’algorithme de Boyer-Moore
RechenmannFrançoisParmentelatThierryVous avez compris que la recherche de motifs, c'est-à-dire de sous-chaînes de caractères dans une chaîne plus importante, était un composant important de beaucoup d'algorithmes de bio-informatique.
-
3.1. Tous les gènes se terminent sur un codon stop
RechenmannFrançoisParmentelatThierryUne fois la séquence d'un génome complet obtenue, débute la phase d'annotation. L'annotation elle-même consiste tout d'abord à rechercher la localisation, c'est-à-dire la position des gènes sur cette
-
3.10. La prédiction de gènes dans les génomes eucaryotes
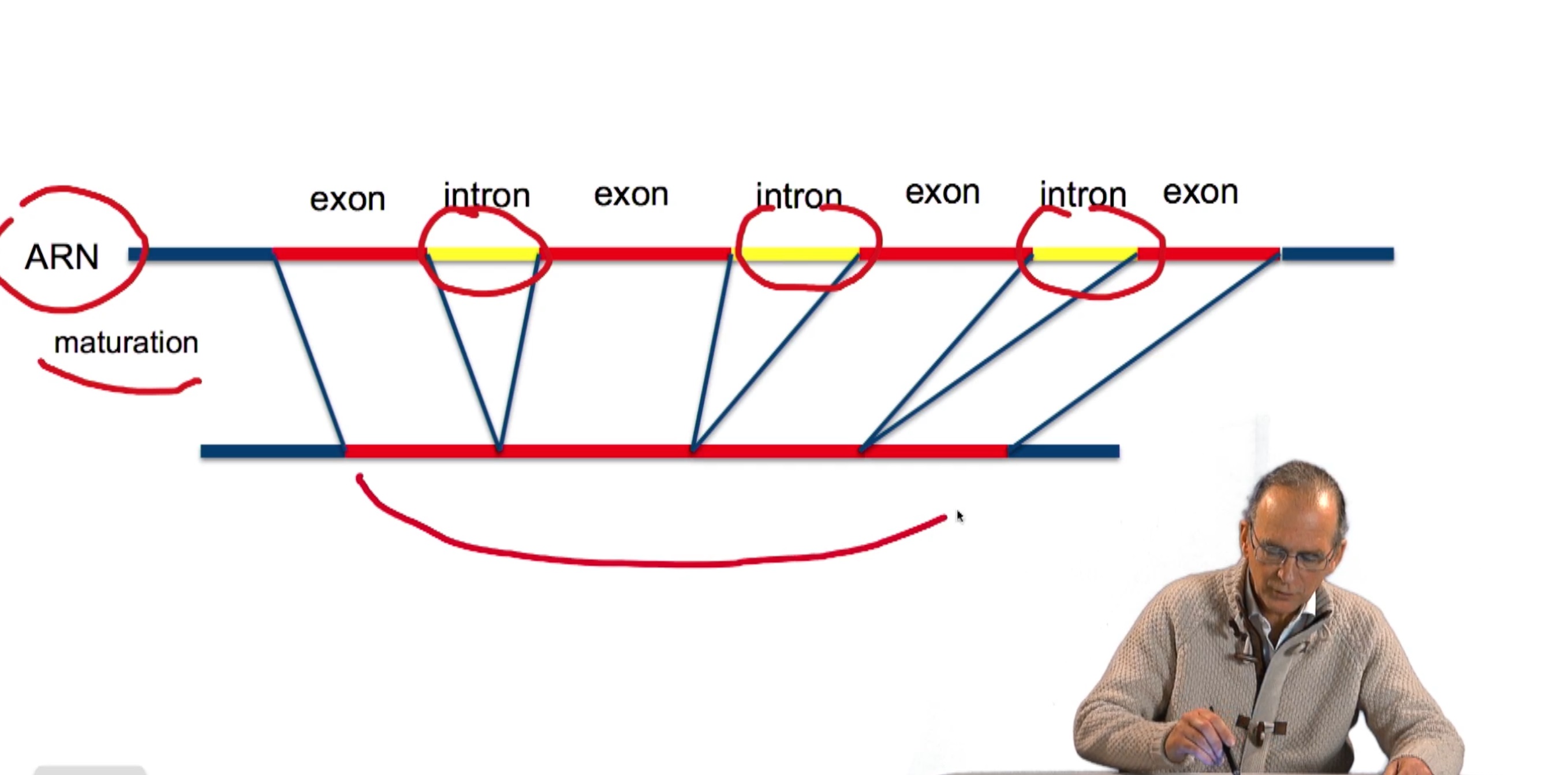
RechenmannFrançoisParmentelatThierrySi nous disposons actuellement de prédicteurs de gènes dans les génomes procaryotes de très bonne efficacité, avec des prédictions relativement fiables, c'est en fait loin d'être le cas sur les
Avec les mêmes intervenants et intervenantes
-
1.2. At the heart of the cell: the DNA macromolecule
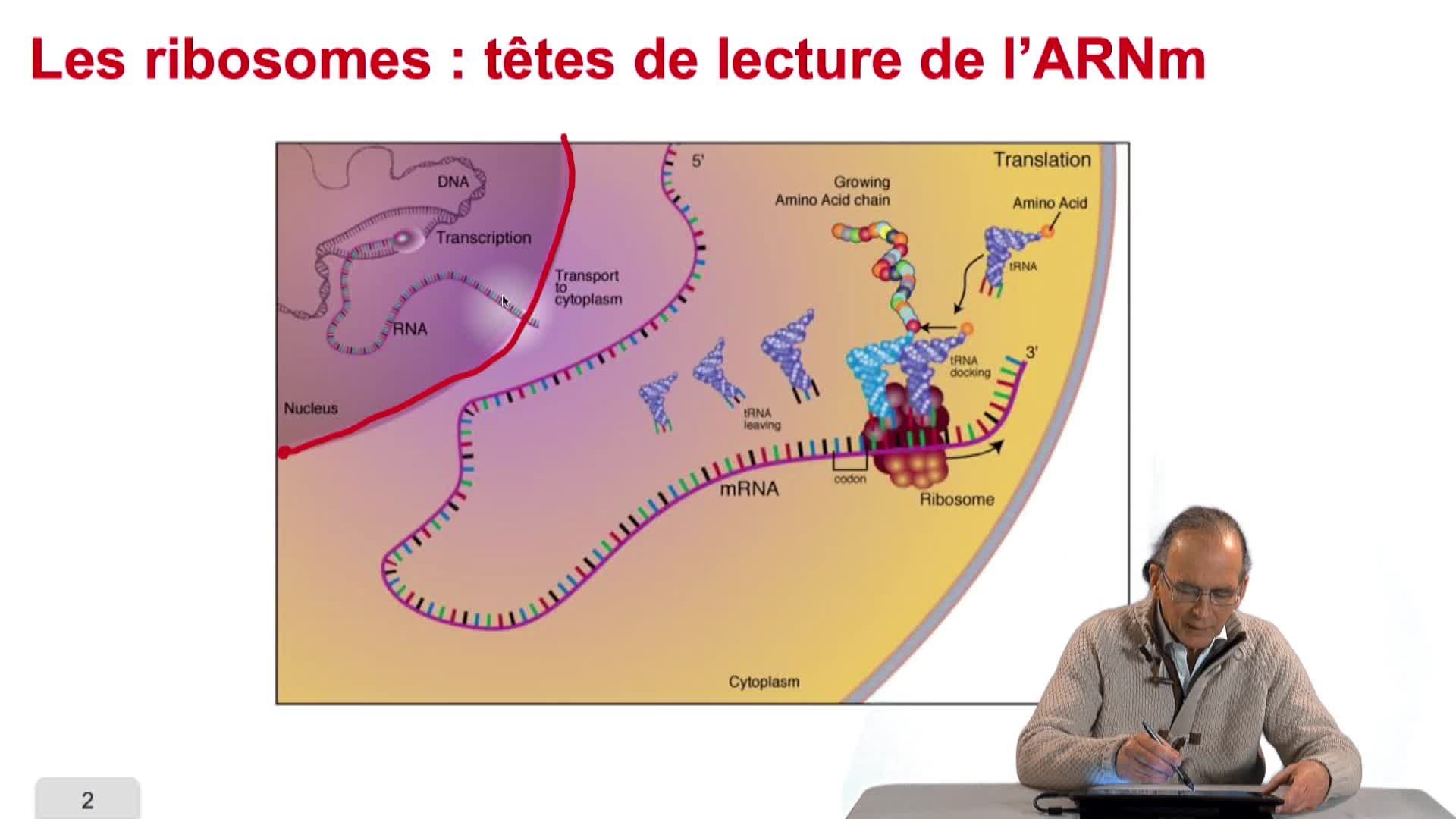
RechenmannFrançoisDuring the last session, we saw how at the heart of the cell there's DNA in the nucleus, sometimes of cells, or directly in the cytoplasm of the bacteria. The DNA is what we call a macromolecule, that
-
1.10. Overlapping sliding window
RechenmannFrançoisWe have made some drawings along a genomic sequence. And we have seen that although the algorithm is quite simple, even if some points of the algorithmare bit trickier than the others, we were able to
-
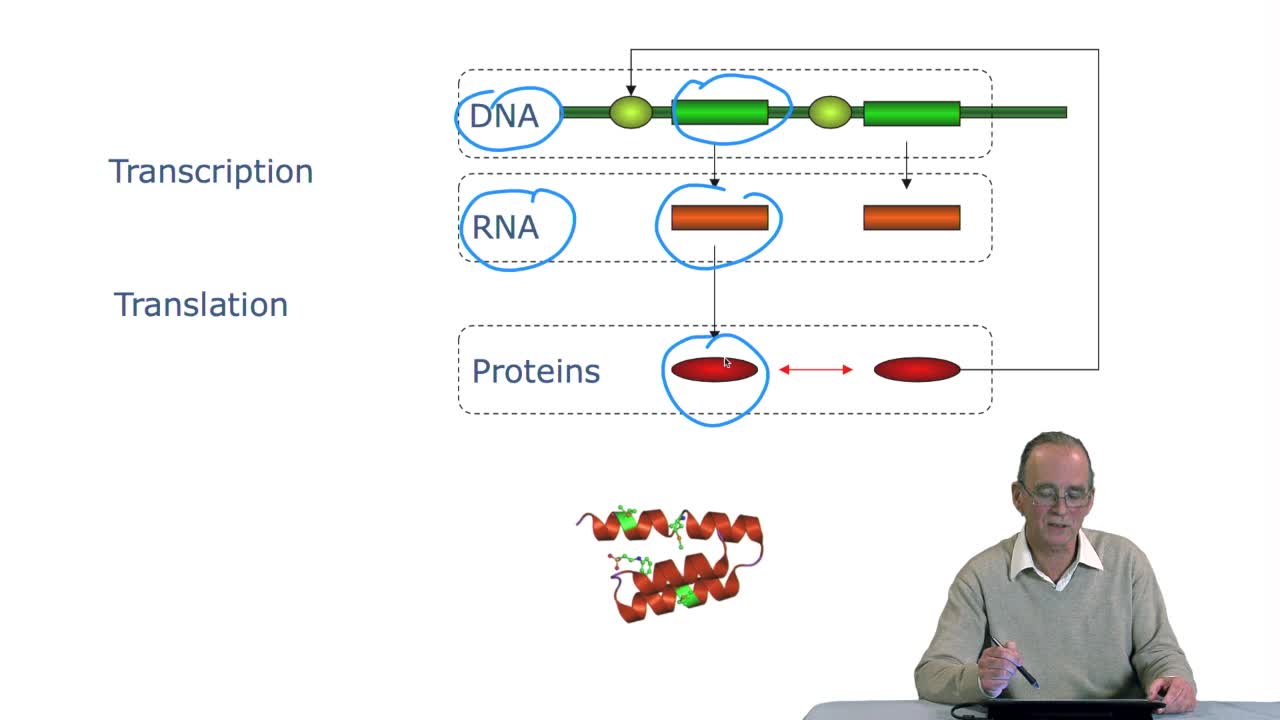
2.3. The genetic code
RechenmannFrançoisGenes code for proteins. What is the correspondence betweenthe genes, DNA sequences, and the structure of proteins? The correspondence isthe genetic code. Proteins have indeedsequences of amino acids.
-
3.6. Boyer-Moore algorithm
RechenmannFrançoisWe have seen how we can make gene predictions more reliable through searching for all the patterns,all the occurrences of patterns. We have seen, for example, howif we locate the RBS, Ribosome
-
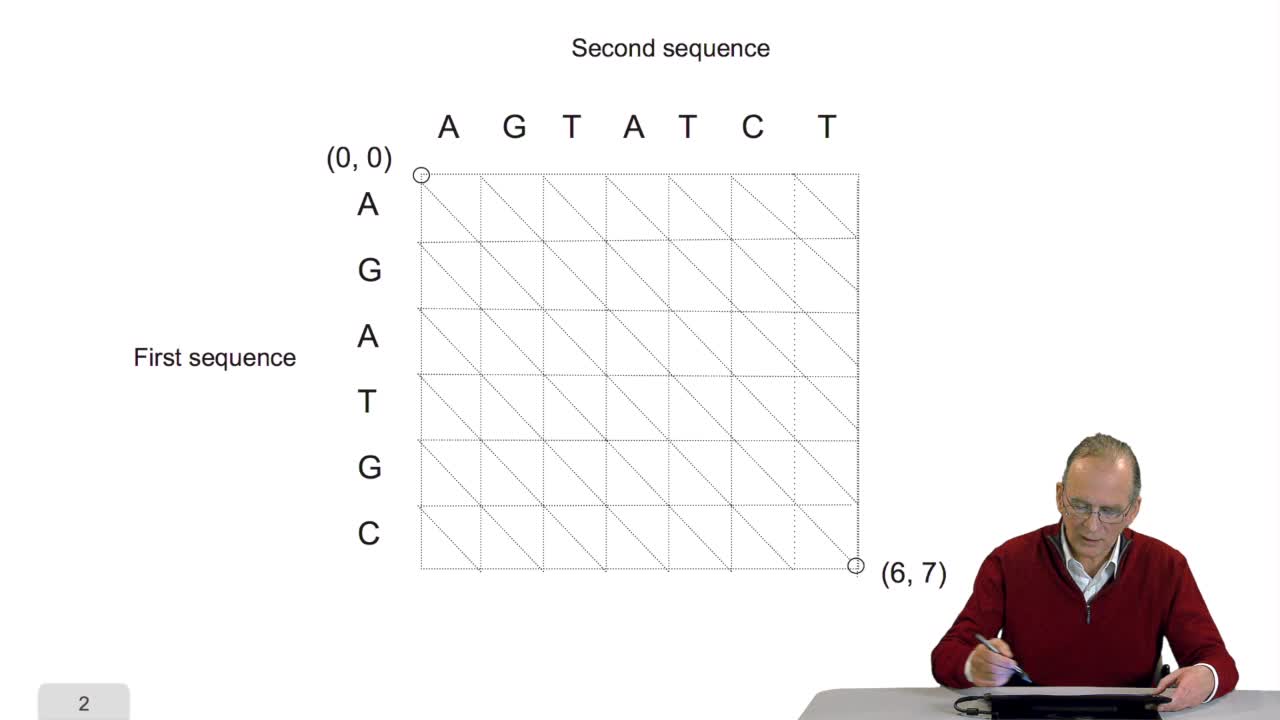
4.5. A sequence alignment as a path
RechenmannFrançoisComparing two sequences and thenmeasuring their similarities is an optimization problem. Why? Because we have seen thatwe have to take into account substitution and deletion. During the alignment, the
-
5.5. Differences are not always what they look like
RechenmannFrançoisThe algorithm we have presented works on an array of distance between sequences. These distances are evaluated on the basis of differences between the sequences. The problem is that behind the
-
1.5. Counting nucleotides
RechenmannFrançoisIn this session, don't panic. We will design our first algorithm. This algorithm is forcounting nucleotides. The idea here is that as an input,you have a sequence of nucleotides, of bases, of letters,
-
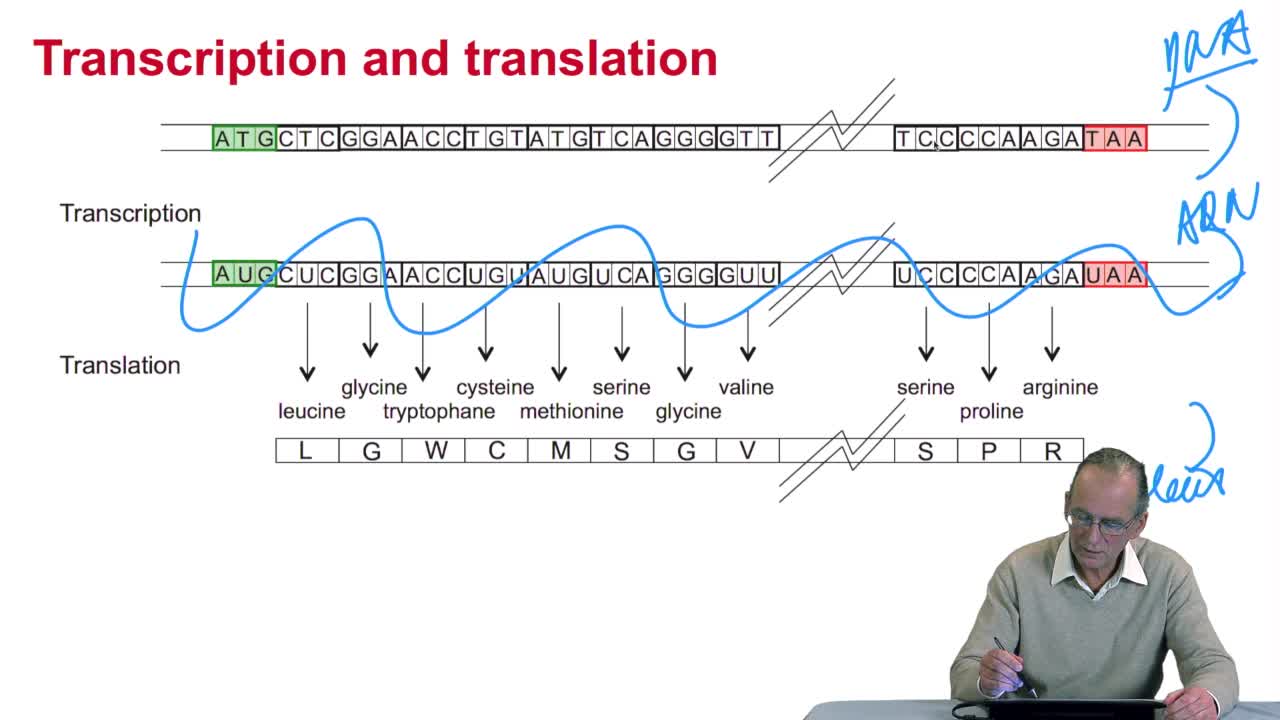
2.4. A translation algorithm
RechenmannFrançoisWe have seen that the genetic codeis a correspondence between the DNA or RNA sequences and aminoacid sequences that is proteins. Our aim here is to design atranslation algorithm, we make the
-
3.1. All genes end on a stop codon
RechenmannFrançoisLast week we studied genes and proteins and so how genes, portions of DNA, are translated into proteins. We also saw the very fast evolutionof the sequencing technology which allows for producing
-
3.9. Benchmarking the prediction methods
RechenmannFrançoisIt is necessary to underline that gene predictors produce predictions. Predictions mean that you have no guarantees that the coding sequences, the coding regions,the genes you get when applying your
-
4.2. Why gene/protein sequences may be similar?
RechenmannFrançoisBefore measuring the similaritybetween the sequences, it's interesting to answer the question: why gene or protein sequences may be similar? It is indeed veryinteresting because the answer is related
-
5.4. The UPGMA algorithm
RechenmannFrançoisWe know how to fill an array with the values of the distances between sequences, pairs of sequences which are available in the file. This array of distances will be the input of our algorithm for
Sur le même thème
-
La voix, une donnée identifiante à protéger
VincentEmmanuelEmmanuel Vincent, chercheur au Centre Inria de l'Université de Lorraine et au Loria (Laboratoire lorrain de recherche en informatique et ses applications), présente sa recherche sur l'anonymisation de
-
Podcast 1/4 d'heure avec : Emmanuel Vincent, chercheur au Centre Inria de l'Université de Lorraine …
VincentEmmanuelRencontre avec Emmanuel Vincent - chercheur au Centre Inria de l'Université de Lorraine et Loria (Laboratoire lorrain de recherche en informatique et ses applications).
-
Stockage de données numériques sur ADN synthétique : Introduction au domaine
AntoniniMarcDuprazElsaLavenierDominiquePrésentation globale des différentes étapes du stockage de données sur des molécules d'ADN synthétique
-
Stockage de données numériques sur ADN synthétique : Production des données: synthèse, séquençage
LavenierDominiqueBarbryPascalDescription des opérations d'écriture et de lecture des molécules d'ADN : synthèse et séquençage.
-
Stockage de données numériques sur ADN synthétique : Reconstruction des données
LavenierDominiqueTraitement des données après séquençage
-
Stockage de données numériques sur ADN synthétique : Codage Canal
DuprazElsaTechniques de codage pour le stockage de données sur ADN
-
Stockage de données numériques sur ADN synthétique : Codage Source
AntoniniMarcCodage source pour le stockage de données sur ADN synthétique
-
Stockage de données numériques sur ADN synthétique : Théorie de l'information
Kas HannaSergeQuelle quantité d'information peut-on stocker et récupérer de manière fiable dans l'ADN ?
-
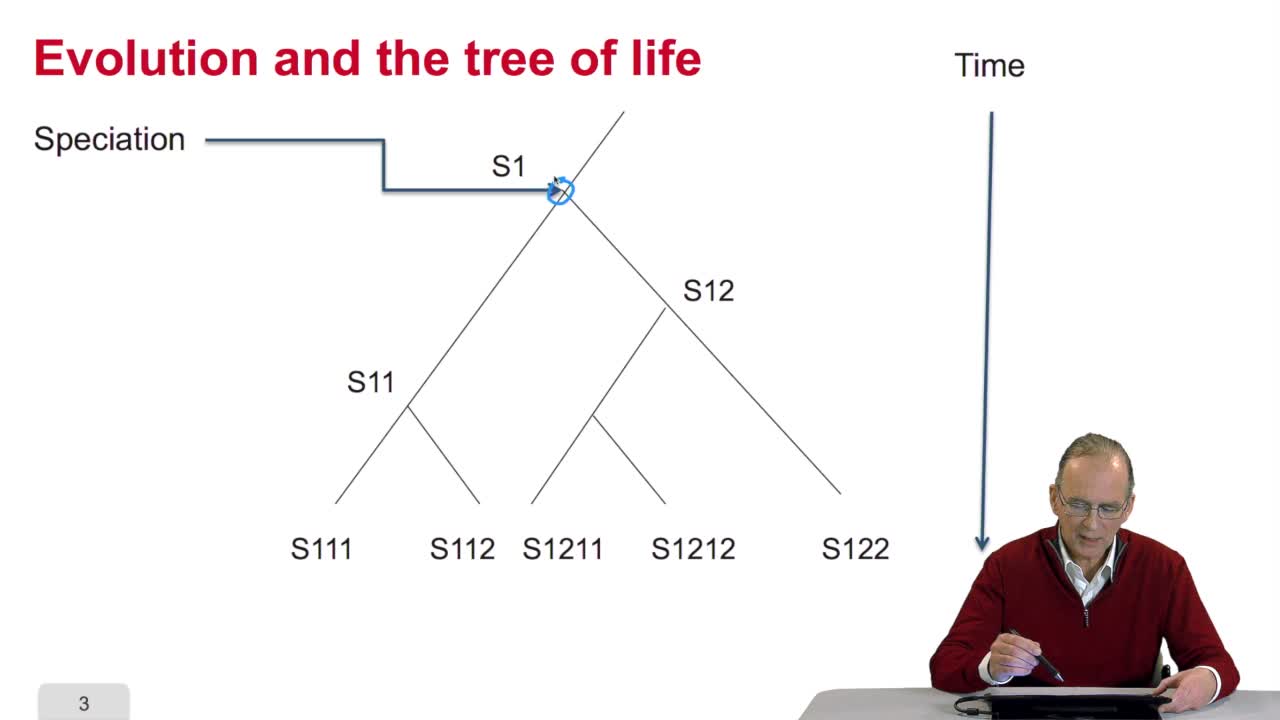
The tree of life
AbbySophieLes Rencontres Exobiologiques pour Doctorants (RED) sont une école de formation sur les « bases de l'astrobiologie ». L’édition 2025 s’est tenue du 16 au 21 mars au Parc Ornithologique du Teich.
-
Machines algorithmiques, mythes et réalités
MazenodVincentVincent Mazenod, informaticien, partage le fruit de ses réflexions sur l'évolution des outils numériques, en lien avec les problématiques de souveraineté, de sécurité et de vie privée...
-
Désassemblons le numérique - #Episode11 : Les algorithmes façonnent-ils notre société ?
SchwartzArnaudLima PillaLaércioEstériePierreSalletFrédéricFerbosAudeRoumanosRayyaChraibi KadoudIkramUn an après le tout premier hackathon sur les méthodologies d'enquêtes journalistiques sur les algorithmes, ce nouvel épisode part à la rencontre de différents points de vue sur les algorithmes.
-
Les machines à enseigner. Du livre à l'IA...
BruillardÉricQue peut-on, que doit-on déléguer à des machines ? C'est l'une des questions explorées par Éric Bruillard qui, du livre aux IA génératives, expose l'évolution des machines à enseigner...