Notice
3.1. Tous les gènes se terminent sur un codon stop
- document 1 document 2 document 3
- niveau 1 niveau 2 niveau 3
Descriptif
Une fois la séquence d'un génome complet obtenue, débute la phase d'annotation. L'annotation elle-même consiste tout d'abord à rechercher la localisation, c'est-à-dire la position des gènes sur cette séquence. Cette semaine, nous allons nous intéresser à la prédiction des gènes, nous allons étudier un algorithme de prédiction de gènes sur des séquences génomiques procaryotes, nous allons essayer d'améliorer la qualité de ces prédictions en ayant recours à des additifs à notre algorithme, recherches de certains motifs supplémentaires, éventuellement recours à des techniques probabilistes. Nous allons chercher à voir comment on peut comparer la qualité des prédictions de différentes méthodes, et à la fin nous étudierons le problème particulier que pose le problème très important de la prédiction des gènes dans un génome eucaryote. Problème encore mal résolu.
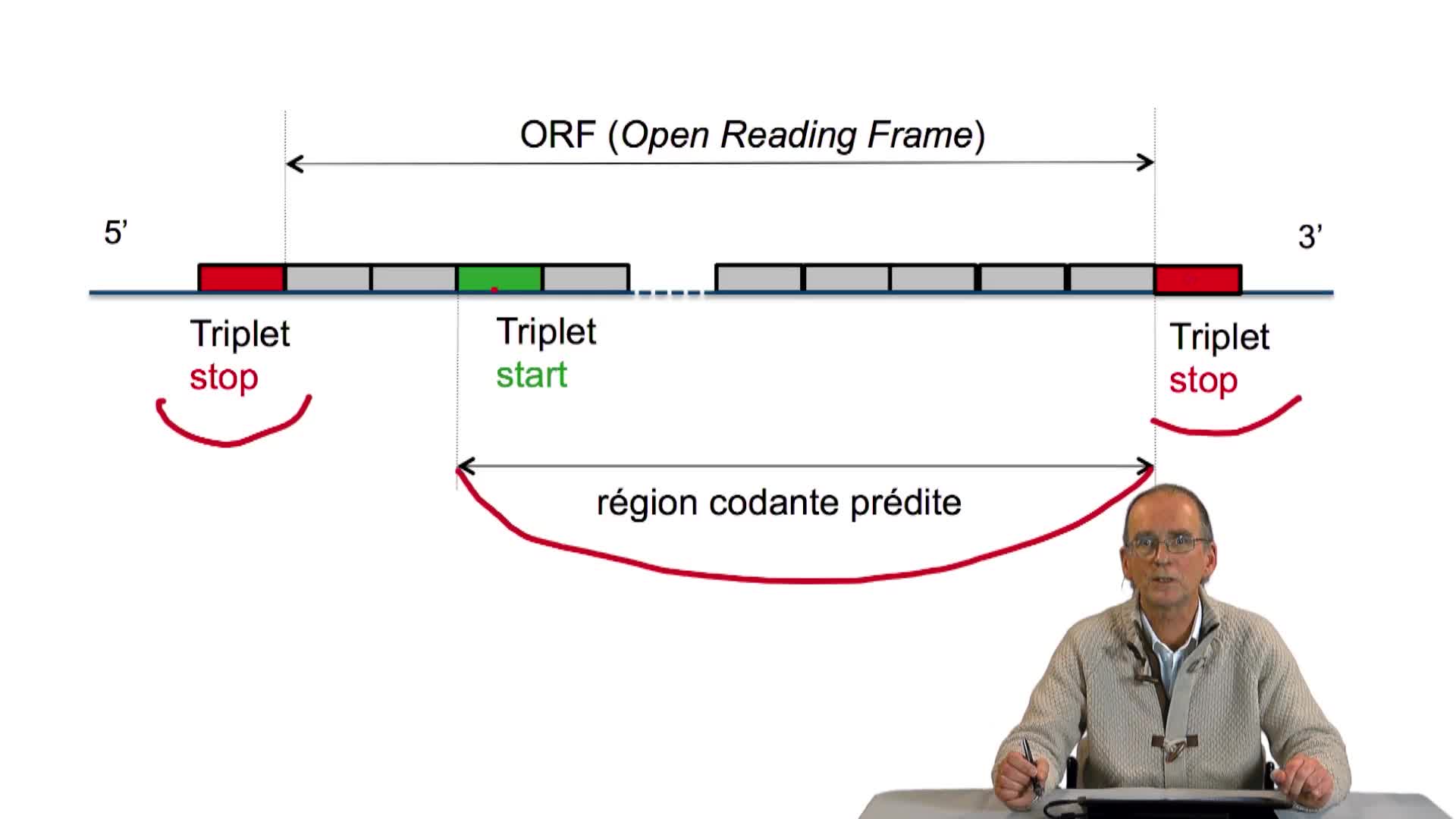
Voilà, pourquoi quand nous parlons ici de prédiction de gènes, nous parlons de prédiction de gènes sur des génomes bactériens. Comment retrouve-t-on des gènes et leurs positions de début et de fin dans un génome bactérien ? Nous connaissons les conditions nécessaires. On sait qu'une région codante, par définition de ce que nous avons vu précédemment, ne peut se situer qu'entre 2 Stop, et des Stop qui sont dans la même phase. 2 Stop consécutifs dans la même phase signifient 2 Stop qui sont séparés par des triplets, autrement dit par un nombre de bases, de nucléotides, de lettres, multiples de 3...
ERRATUM
Slide 6 : La marque de fin d'ORF est incorrectement positionnée sur la slide 6. En effet, le triplet STOP ne fait pas partie de l'ORF, même s'il la délimite. La slide 7 est correcte.
Intervention
Dans la même collection
-
3.7. Index et arbre des suffixes
RechenmannFrançoisParmentelatThierryIl y a donc deux approches pour améliorer la performance des algorithmes de recherche d'un motif dans une chaîne de caractères. La première approche consiste à pré-traiter le motif. On a vu un exemple
-
3.2. Un algorithme simple de prédiction de gènes
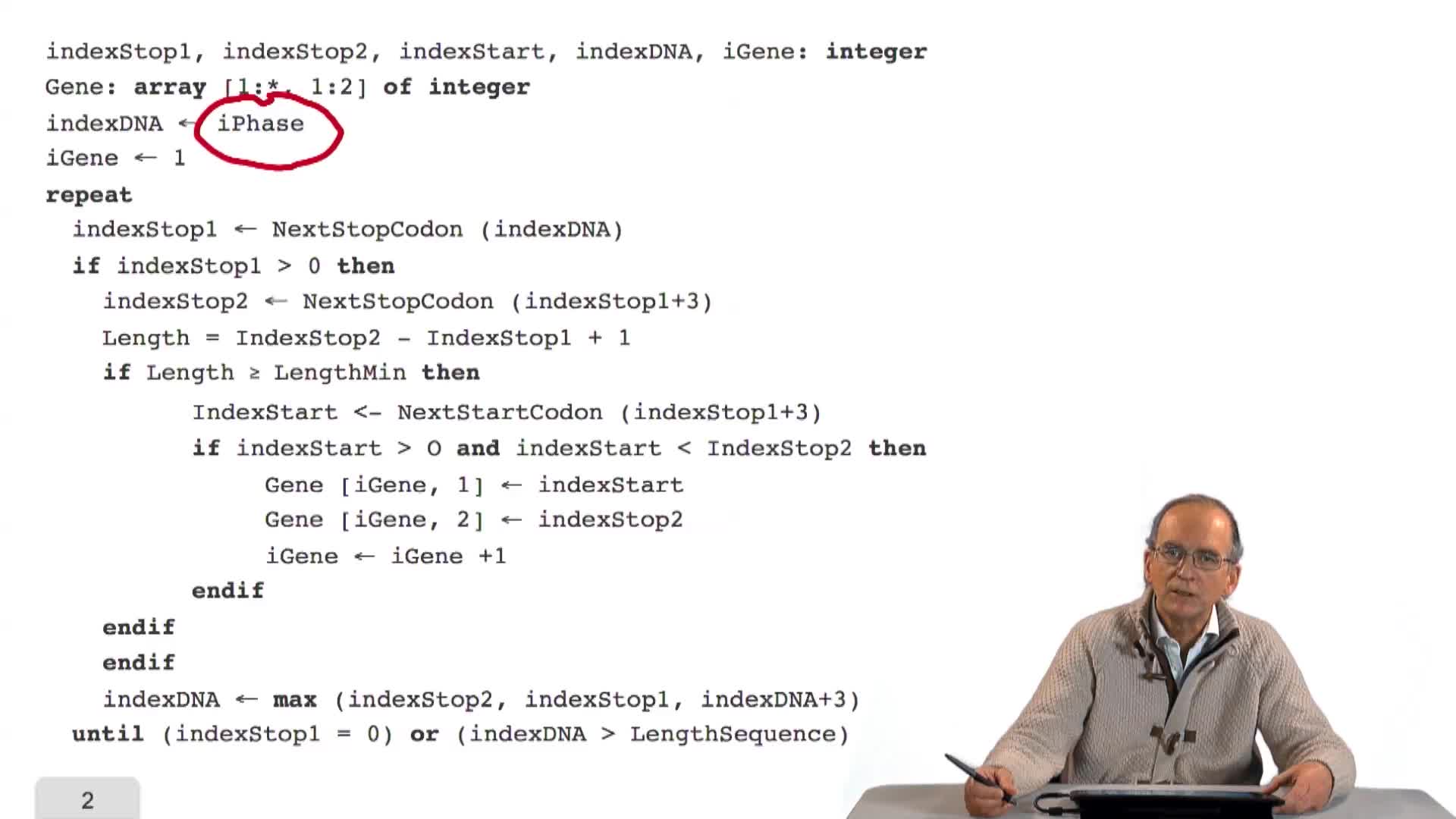
RechenmannFrançoisParmentelatThierrySur la base des principes énoncés précédemment, nous allons écrire un premier algorithme de prédiction de gènes sur un texte génomique procaryote. Je rappelle ces principes. L'idée est la suivante :
-
3.10. La prédiction de gènes dans les génomes eucaryotes
RechenmannFrançoisParmentelatThierrySi nous disposons actuellement de prédicteurs de gènes dans les génomes procaryotes de très bonne efficacité, avec des prédictions relativement fiables, c'est en fait loin d'être le cas sur les
-
3.5. Comment améliorer la qualité des prédictions ?
RechenmannFrançoisParmentelatThierryIl faut toujours le répéter et le souligner, les algorithmes qui déterminent des gènes déterminent des gènes candidats. Ce sont des prédictions de gènes. Donc la question est de savoir s'il est
-
3.8. Des méthodes probabilistes à la rescousse
RechenmannFrançoisParmentelatThierryNous avons vu comment la qualité des prédictions de gènes dans un génome bactérien, pouvait être améliorée à travers la recherche d'occurrences de motifs particuliers liés au site de fixation du
-
3.3. À la recherche des codons start et stop
RechenmannFrançoisParmentelatThierryNous avons écrit la structure, l'ossature d'un algorithme de prédiction de gènes dans un génome bactérien, en utilisant les principes que nous avions énoncés précédemment. Cet algorithme est incomplet
-
3.6. L’algorithme de Boyer-Moore
RechenmannFrançoisParmentelatThierryVous avez compris que la recherche de motifs, c'est-à-dire de sous-chaînes de caractères dans une chaîne plus importante, était un composant important de beaucoup d'algorithmes de bio-informatique.
-
3.9. Comment évaluer la qualité de prédiction des méthodes ?
RechenmannFrançoisParmentelatThierryNous avons vu qu'il était possible, ou du moins nous le pensions, améliorer la qualité de prédiction des gènes sur un génome bactérien en introduisant des démarches supplémentaires, de recherches de
-
3.4. Prédiction de tous les gènes d’une séquence
RechenmannFrançoisParmentelatThierryEn combinant de façon adéquate la recherche des triplés Stop et Start sur un brin d'ADN, nous avons obtenu un algorithme qui prédit les gènes sur ce brin, mais également sur une phase. C'est-à-dire en
Avec les mêmes intervenants et intervenantes
-
1.7. DNA walk
RechenmannFrançoisWe will now design a more graphical algorithm which is called "the DNA walk". We shall see what does it mean "DNA walk". Walk on to DNA. Something like that, yes. But first, just have a look again at
-
2.6. Algorithms + data structures = programs
RechenmannFrançoisBy writing the Lookup GeneticCode Function, we completed our translation algorithm. So we may ask the question about the algorithm, does it terminate? Andthe answer is yes, obviously. Is it pertinent,
-
3.3. Searching for start and stop codons
RechenmannFrançoisWe have written an algorithm for finding genes. But you remember that we arestill to write the two functions for finding the next stop codonand the next start codon. Let's see how we can do that. We
-
4.1. How to predict gene/protein functions?
RechenmannFrançoisLast week we have seen that annotating a genome means first locating the genes on the DNA sequences that is the genes, the region coding for proteins. But this is indeed the first step,the next very
-
4.10. How efficient is this algorithm?
RechenmannFrançoisWe have seen the principle of an iterative algorithm in two paths for aligning and comparing two sequences of characters, here DNA sequences. And we understoodwhy the iterative version is much more
-
5.7. The application domains in microbiology
RechenmannFrançoisBioinformatics relies on many domains of mathematics and computer science. Of course, algorithms themselves on character strings are important in bioinformatics, we have seen them. Algorithms and
-
1.2. At the heart of the cell: the DNA macromolecule
RechenmannFrançoisDuring the last session, we saw how at the heart of the cell there's DNA in the nucleus, sometimes of cells, or directly in the cytoplasm of the bacteria. The DNA is what we call a macromolecule, that
-
1.10. Overlapping sliding window
RechenmannFrançoisWe have made some drawings along a genomic sequence. And we have seen that although the algorithm is quite simple, even if some points of the algorithmare bit trickier than the others, we were able to
-
2.3. The genetic code
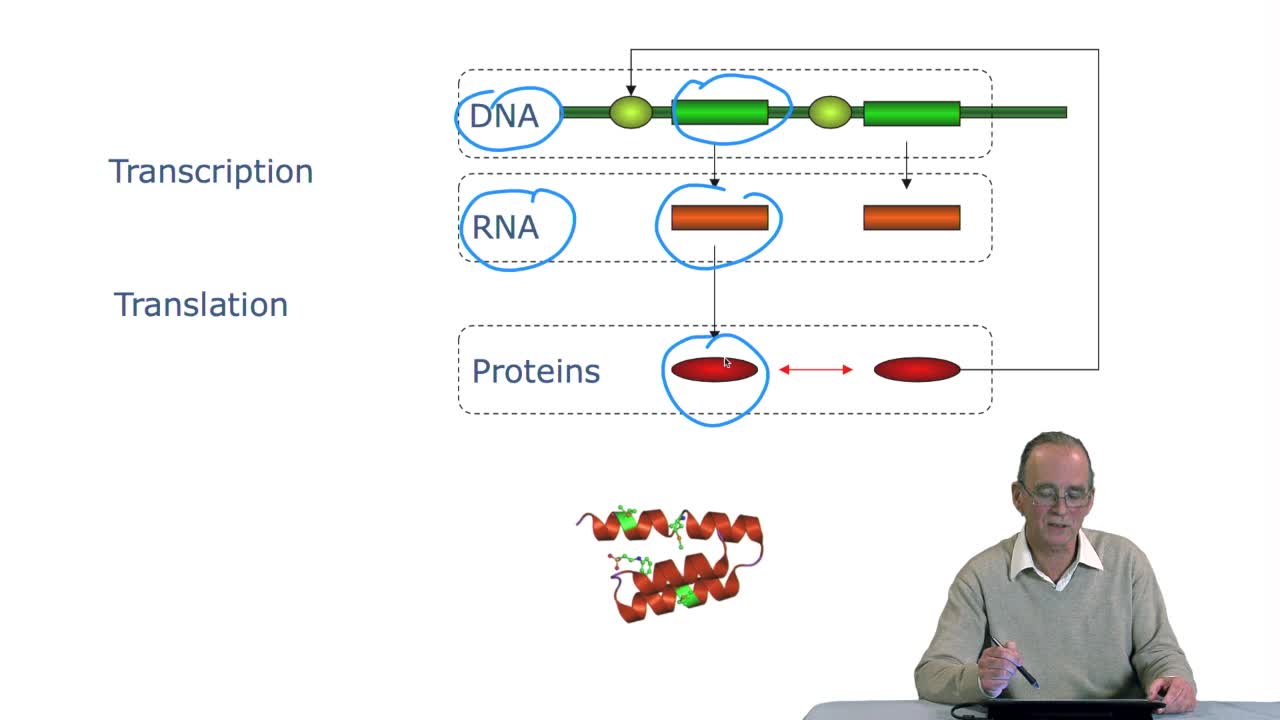
RechenmannFrançoisGenes code for proteins. What is the correspondence betweenthe genes, DNA sequences, and the structure of proteins? The correspondence isthe genetic code. Proteins have indeedsequences of amino acids.
-
3.6. Boyer-Moore algorithm
RechenmannFrançoisWe have seen how we can make gene predictions more reliable through searching for all the patterns,all the occurrences of patterns. We have seen, for example, howif we locate the RBS, Ribosome
-
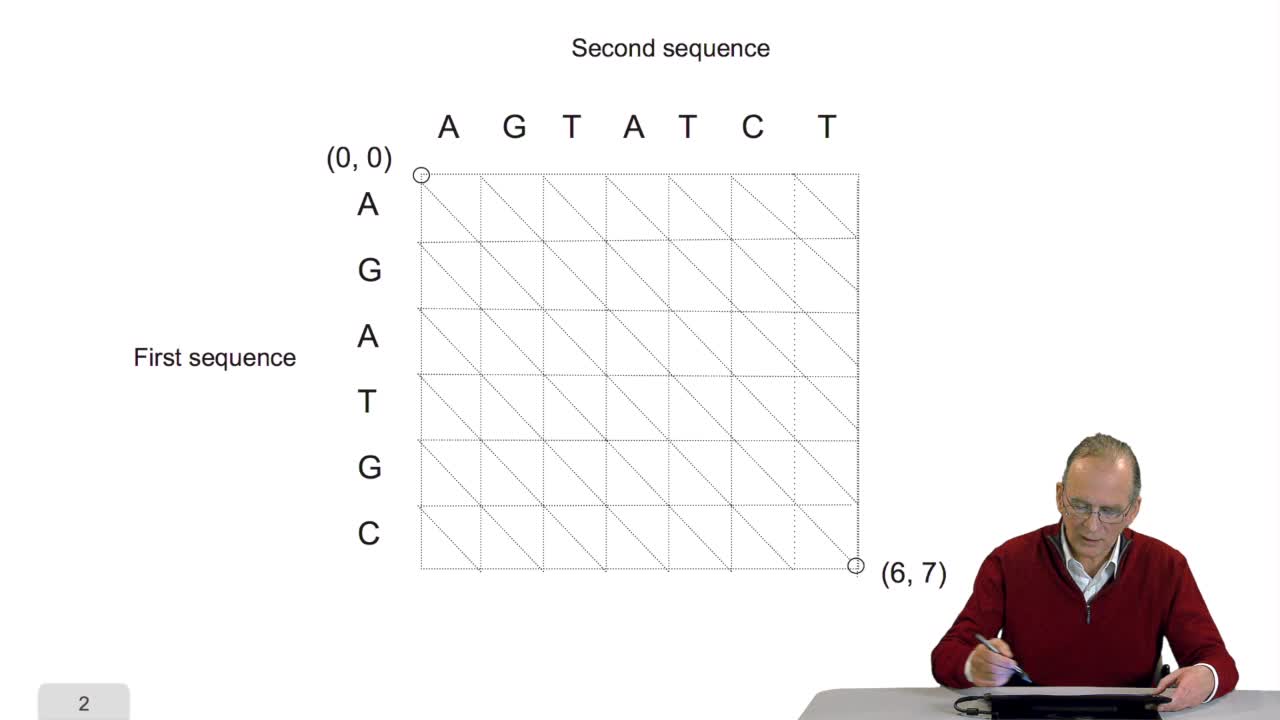
4.5. A sequence alignment as a path
RechenmannFrançoisComparing two sequences and thenmeasuring their similarities is an optimization problem. Why? Because we have seen thatwe have to take into account substitution and deletion. During the alignment, the
-
5.5. Differences are not always what they look like
RechenmannFrançoisThe algorithm we have presented works on an array of distance between sequences. These distances are evaluated on the basis of differences between the sequences. The problem is that behind the
Sur le même thème
-
The tree of life
AbbySophieLes Rencontres Exobiologiques pour Doctorants (RED) sont une école de formation sur les « bases de l'astrobiologie ». L’édition 2025 s’est tenue du 16 au 21 mars au Parc Ornithologique du Teich.
-
Machines algorithmiques, mythes et réalités
MazenodVincentVincent Mazenod, informaticien, partage le fruit de ses réflexions sur l'évolution des outils numériques, en lien avec les problématiques de souveraineté, de sécurité et de vie privée...
-
Désassemblons le numérique - #Episode11 : Les algorithmes façonnent-ils notre société ?
SchwartzArnaudLima PillaLaércioEstériePierreSalletFrédéricFerbosAudeRoumanosRayyaChraibi KadoudIkramUn an après le tout premier hackathon sur les méthodologies d'enquêtes journalistiques sur les algorithmes, ce nouvel épisode part à la rencontre de différents points de vue sur les algorithmes.
-
Les machines à enseigner. Du livre à l'IA...
BruillardÉricQue peut-on, que doit-on déléguer à des machines ? C'est l'une des questions explorées par Éric Bruillard qui, du livre aux IA génératives, expose l'évolution des machines à enseigner...
-
Désassemblons le numérique - #Episode9 : Bientôt des supercalculateurs dans nos piscines ?
BeaumontOlivierBouzelRémiDes supercalculateurs feraient-ils bientôt leur apparition dans les piscines municipales pour les chauffer ? Réponses d'Olivier Beaumont, responsable de l'équipe-projet Topal, et Rémi Bouzel,
-
Le projet dnarXiv : Stockage de données sur des molécules d'ADN
LavenierDominiqueDuprazElsaLeblancJulienCoatrieuxGouenouDominique Lavenier, Elsa Dupraz, Julien Leblanc et Gouenou Coatrieux nous présentent le projet dnarXiv, un projet porté par le LabEx CominLabs qui explore le stockage de données sur des molécules d
-
Projection methods for community detection in complex networks
LitvakNellyCommunity detection is one of most prominent tasks in the analysis of complex networks such as social networks, biological networks, and the world wide web. A community is loosely defined as a group
-
Lara Croft. doing fieldwork under surveillance
Dall'AgnolaJasminLara Croft. Doing Fieldwork Under Surveillance Intervention de Jasmin Dall'Agnola (The George Washington University), dans le cadre du Colloque coorganisé par Anders Albrechtslund, professeur en
-
Containing predictive tokens in the EU
CzarnockiJanContaining Predictive Tokens in the EU – Mapping the Laws Against Digital Surveillance, intervention de Jan Czarnocki (KU Leuven), dans le cadre du Colloque coorganisé par Anders Albrechtslund,
-
Ivan Murit - Processus de création d'images
MuritIvanJe vais présenter une manière décalée d'aborder les outils d'impression. Pour cela nous ne partirons pas de l'envie d'imprimer une image préexistante, mais d'avant cela : comment se crée une forme
-
Le Creativ’Lab, au cœur de la robotique et de l’intelligence artificielle (ASR N°18 - LORIA)
HénaffPatrickLefebvreSylvainLe LORIA, laboratoire phare de la Grande Région dans le domaine de l’informatique, propose de rendre la recherche plus ouverte, plus collaborative, plus ambitieuse… en un mot, plus créative, à travers
-
Les algorithmes de Parcoursup
MathieuClaireL’objectif de la journée « Algorithmes d’aide à la décision publique » était de sensibiliser le grand public aux rôles des algorithmes d’aide à la décision publique utilisés par exemple pour l